泛素
此條目需要精通或熟悉相關主題的編者參與及協助編輯。 (2016年3月6日) |
泛素(英語:ubiquitin)又稱泛素蛋白[1]、泛蛋白[2],是一種存在於大多數真核細胞中高度保守的熱穩定小分子基因表達調控蛋白;因廣泛存在於真核細胞中而得名。 它於1975年由吉迪恩·格魯斯坦(Gideon Goldstein)發現,並在1970年代末和1980年代進一步定性。[3] 人類基因組中有四個泛素基因: UBB、UBC、UBA52 和 RPS27A。
| Ubiquitin family | |
|---|---|
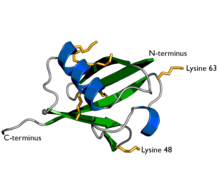 泛素示意圖 。7個賴氨酸側鏈以黃色/橙色顯示。 | |
| 鑑定 | |
| 標誌 | 泛素 (ubiquitin) |
| InterPro | IPR000626 |
| PROSITE | PDOC00271 |
| SCOP | 1aar / SUPFAM |
泛素使用泛素連接酶與其他蛋白質進行酶促偶聯,其特性因這種泛素化而改變。根據泛素鍵的數量和類型,泛素化靶蛋白與其他蛋白質的相互作用可以被促進或阻礙,其活性可以受到影響,其在細胞中的定位可以被改變,或者可以加速其降解。在蛋白質質量控制過程中,幾個連接成鏈的泛素標記多泛素化蛋白質,以便在蛋白酶體中降解。泛素化對於轉錄和翻譯的調節也很重要,參與信號轉導和胞吞作用,參與DNA修復,並發生在細胞周期、細胞分化和炎症反應的調節過程中。
在底物蛋白質上添加泛素稱為泛素化(ubiquitylation)。泛素化對蛋白質有多種影響:它可以標記蛋白質,使其通過蛋白酶體降解,改變其在細胞中的位置,影響其活性,促進或阻止蛋白質交互作用。泛素化包括三個主要步驟:激活、結合和連接,分別由泛素活化酶(E1s)、泛素綴合酶(E2s)和泛素連接酶(E3s)完成。這一連續級聯的結果是,泛素通過異肽鍵與蛋白質底物上的離胺酸 (Lysine)殘基結合,通過硫酯鍵與半胱氨酸殘基結合,通過硫酯鍵與絲氨酸和蘇氨酸殘基結合,或通過肽鍵與蛋白質N端的氨基結合。
蛋白質修飾可以是單個泛素蛋白(單泛素化),也可以是泛素鏈(多泛素化)。次級泛素分子總是與前一個泛素分子的七個賴氨酸殘基或N端甲硫氨酸中的一個相連。
與此相反,有一系列去泛素化酶(DUB)可以催化裂解已經附着的泛素分子等。
泛素化代表蛋白質的翻譯後修飾。類似的修飾是泛素樣蛋白質的偶聯,例如SUMO、Urm1 或 NEDD8,相應地稱為 sumoylation、urmylation 或 neddylation。此外,在一些原核生物中,例如結核分枝桿菌中,已知有一種類似於泛素的蛋白質,稱為原核泛素樣蛋白(Prokaryotic ubiquitin-like protein, Pup)。
鑑定
編輯泛素(原名稱為泛在免疫多肽)於1975年首次被發現[6] ,是一種在所有真核細胞中表達的 8.6 kDa 蛋白質。1980年代初,阿龍·切哈諾沃(Aaron Chicanover)、歐文·羅斯(Irwin Rose)和阿夫拉姆·赫什科(Abram Hershko)在以色列理工學院闡明了泛素的基本功能和泛素化途徑的組成部分,並因此於2004年獲得諾貝爾化學獎。[4]
蛋白質
編輯| 殘基數量 | 76 |
|---|---|
| 分子量 | 8564.8448 Da |
| 等電點 (pI) | 6.79 |
| 基因名稱 | RPS27A (UBA80, UBCEP1), UBA52 (UBCEP2), UBB, UBC |
| 序列 (單字母符號) | MQIFVKTLTGKTITLEVEPSDTIENVKAKIQDKEGIPPD QQRLIFAGKQLEDGRTLSDYNIQKESTLHLVLRLRGG |
泛素是一種存在於所有真核細胞中的小型蛋白質。它通過與大量目標蛋白質結合來實現其多種功能。泛素可發生多種不同的修飾。泛素蛋白本身由 76個氨基酸組成,分子質量約為 8.6 kDa (道爾頓)。其主要特徵包括C端尾部和7個賴氨酸殘基。它在整個真核生物進化過程中高度保守;人類的和酵母的泛素的序列相同度高達96%。[7]
基因
編輯哺乳動物體內的泛素由四種不同的基因編碼。UBA52和RPS27A基因分別編碼一個與核糖體蛋白L40和S27a融合的泛素拷貝。UBB和UBC基因編碼多泛素前體蛋白。[8]
泛素化
編輯泛素化是一種酶促的、蛋白質翻譯後修飾(PTM)過程,其中來自被激活泛素的二甘氨酸模體中末端甘氨酸的羧基與被修飾蛋白中賴氨酸ε氨基相連形成一酰胺鍵。
將蛋白標記上泛素的過程(泛素化)包括了以下一系列步驟:
- 活化:泛素通過泛素活化酶 E1 參與的一個兩步反應而被激活,並且需要 ATP 提供能量。 第一步是泛素與ATP反應,形成泛素-腺苷酸複合物; 第二步是泛素的轉移,即泛素與 AMP 分離並轉移到 E1 的半胱氨酸殘基上,泛素的羧基端與 E1 酶中半胱氨酸的巰基通過硫酯鍵相連。
- 結合:E1將活化後的泛素通過轉硫酯化反應轉移到泛素結合酶E2。
- 連接:泛素連接酶 E3 催化泛素級聯反應的最後一步,將結合到 E2 的泛素轉移到目標蛋白上,使目標蛋白的一個賴氨酸與泛素的羧基端的一個甘氨酸通過異構肽鍵連接,通常這個步驟需要100多個E3連接酶的催化, E3 連接酶具有底物識別作用,並且能夠與 E2 和底物相作用,有些 E3 也可以激活 E2 的活性。
在泛素化級聯反應中各個步驟分為不同的層次的, E1 能夠結合多個 E2, E2 能夠結合多個E3,這種嚴密的分層現象能夠使細胞對泛素化進行更為精緻的調節。
泛素樣蛋白 ubiquitin-like proteins(ULPs)也通過 E1–E2–E3 級聯繫統對蛋白質進行修飾,不過與泛素蛋白的修飾級聯繫統也存在一些差異。
E4 泛素鏈延長酶, 能夠在 E3 泛素連接酶產生的單泛素化蛋白上延長泛素。
E3
編輯E3泛素連接酶具有兩個結構域中的一個:
- HECT(Homologous to the E6-AP Carboxyl Terminus)結構域
- RING(Really Interesting New Gene)結構域(or the closely related U-box domain)
泛素最終通過兩種方式轉移至蛋白質底物:
- 直接從E2結合酶轉移至蛋白質底物(由E3酶的 RING 結構域催化)
- 通過E3連接酶再轉移至蛋白質底物(由E3酶的 HECT 結構域催化),在轉移至蛋白質底物之前,泛素與E3酶 HECT 結構域中保守的半胱氨酸殘基通過硫酯鍵而形成E3-泛素複合物。
去泛素化
編輯蛋白質的泛素化是一個可逆的過程,這一過程叫做去泛素化(deubiquitination),由一類蛋白水解酶——去泛素化酶(deubiquitination enzymes,DUBs)介導。[9]
疾病關聯
編輯發病機制
編輯泛素通路與多種疾病和病症的發病機制有關,包括:[10]
神經退行性疾病
編輯泛素與蛋白穩態功能障礙相關的神經退行性疾病有關,包括阿爾茨海默病、運動神經元疾病[11]、亨丁頓舞蹈症和帕金森氏病。在與阿爾茨海默病和帕金森病相關的病變中發現了編碼泛素-1不同異構體的轉錄本變體。[12]研究表明,大腦中泛素水平較高可減少前類澱粉蛋白質 (APP) 的畸形,而前類澱粉蛋白質 (APP) 是引發阿爾茨海默病的關鍵因素。[13] 相反,大腦中泛素-1 水平較低則與 APP 畸形增加有關。[13] 泛素B的移碼突變可導致截短肽缺失C端甘氨酸。這種異常肽被稱為 UBB+1,已被證明會在阿爾茨海默病和其他 tauopathies 中選擇性積累。
感染與免疫
編輯泛素和泛素樣分子廣泛調節免疫信號轉導途徑的幾乎所有階段,包括穩態抑制、感染期間的激活和清除後的衰減。如果沒有這種調節,針對病原體的免疫激活可能會出現缺陷,導致慢性疾病或死亡。或者,免疫系統可能過度激活,器官和組織可能受到自體免疫損傷。
另一方面,病毒必須阻斷或重定向宿主細胞過程(包括免疫)才能有效複製,但許多與疾病相關的病毒的基因組信息有限。由於泛素系統在細胞中的作用非常多,因此操縱泛素系統是此類病毒阻斷、顛覆或重定向宿主細胞關鍵過程以支持自身複製的有效方法。[14]
維甲酸誘導基因-I[ (RIG-I) 蛋白是人體細胞中病毒和其他侵入性 RNA 的主要免疫系統傳感器。[15] RIG-I 樣受體 (RLR) 免疫信號通路是泛素在免疫調節中的作用方面研究最廣泛的通路之一。[16]
遺傳性疾病
編輯- 安格曼症候群是由 UBE3A 的破壞引起的,UBE3A 編碼一種稱為 E6-AP 的泛素連接酶 (E3)。
- 希佩爾-林道綜合徵涉及一種稱為 VHL 腫瘤抑制基因或 VHL 基因的泛素 E3 連接酶的破壞。
- 范可尼貧血症:十三個已知基因中,有八個基因的破壞可導致這種疾病,這些基因編碼的蛋白質會形成大型泛素連接酶 (E3) 複合物。
- 3-M綜合徵是一種常染色體隱性生長遲緩症,與 Cullin7 E3 泛素連接酶突變有關。[17]
診斷用途
編輯使用泛素抗體進行免疫組化可識別細胞內該蛋白質的異常積累,從而提示疾病過程。這些蛋白質堆積被稱為包涵體(指細胞中任何微觀可見的異常物質聚集的統稱)。示例包括:
- 阿爾茨海默病中的神經原纖維纏結(Neurofibrillary tangle)
- 帕金森病中的路易氏體 (Lewy body)
- 額顳葉失智症 (最初被稱為皮克病) 中的皮克小體 (Pick body)
- 運動神經元病和亨廷頓病中的包涵體
- 酒精性肝病中的麥洛利氏小體 (Mallory body)
- 星形膠質細胞中的羅森塔爾纖維 (Rosenthal fiber)
與癌症的聯繫
編輯蛋白質的翻譯後修飾是真核細胞[訊息傳遞 (生物)|信號傳導]]過程中普遍採用的一種機制。[18] 泛素化,即蛋白質的泛素連接,是細胞周期進展和細胞增殖發育的一個關鍵過程。 雖然泛素化通常作為通過26S蛋白酶體進行蛋白質降解的信號,但它也可以用於其他基本細胞過程,如胞吞作用[19]、酶激活[20]和DNA修復。[21]此外,由於泛素化嚴格調節細胞周期蛋白的細胞水平,其失調預計會產生嚴重影響。由於蛋白酶體抑制劑具有高抗腫瘤活性,首次觀察到泛素/蛋白酶體通路在致癌過程中的重要性的證據。[22][23][24]各種研究表明,泛素化過程的缺陷或改變通常與人類癌症有關或存在於人類癌症中。[25][26][27][28][29][30][31][32]惡性腫瘤可能通過腫瘤抑制基因的功能喪失突變、泛素化活性增加和/或由於相關蛋白質的突變導致泛素化的間接減弱而發展。[33]
E3泛素連接酶直接功能缺失突變
編輯腎細胞癌
編輯VHL(Von Hippel–Lindau)基因編碼E3泛素連接酶的組成部分。VHL複合物靶向缺氧誘導轉錄因子家族 (HIF) 的一個成員,通過在常氧條件下與氧依賴性破壞域相互作用進行降解。HIF激活下游靶標,如血管內皮生長因子 (VEGF),促進血管新生。VHL突變會阻止HIF降解,從而導致血管過多病變和腎腫瘤的形成。
乳腺癌
編輯BRCA1基因是人類的另一種腫瘤抑制基因,它編碼參與對DNA損傷反應的BRCA1蛋白。該蛋白質含有具有E3泛素連接酶活性的RING基序。BRCA1可以與其他分子(如BARD1和BAP1)形成二聚體,以發揮其泛素化活性。經常發現影響連接酶功能的突變,並且與各種癌症有關。
細胞周期蛋白E
編輯由於細胞周期進程是細胞生長和分化的最基本過程,並且是人類癌症中最常見的改變,因此預計細胞周期調節蛋白會受到嚴格調控。顧名思義,細胞周期蛋白的水平僅在細胞周期的某個時間點較高。這是通過泛素化和降解持續控制細胞周期蛋白或CDK水平來實現的。當細胞周期蛋白E與CDK2結合併被磷酸化時,SCF相關F-box蛋白Fbw7會識別該複合物並將其作為降解目標。超過30%的人類腫瘤中發現了Fbw7突變,這使其成為一種腫瘤抑制蛋白。[32]
磷酸化依賴性泛素化
編輯泛素化和磷酸化之間的相互作用一直是人們研究的熱點,因為磷酸化通常作為泛素化導致降解的標誌。 此外,泛素化還可以起到打開/關閉蛋白質激酶活性的作用。[34] 磷酸化的關鍵作用主要體現在激活和去除Cbl蛋白的自抑制作用。[35] Cbl是一種 E3泛素連接酶,其RING指結構域與其酪氨酸激酶結合 (TKB) 結構域相互作用,從而阻止RING結構域與E2泛素結合酶相互作用。這種分子內相互作用是一種自抑制調節,可阻止其作為各種生長因子和酪氨酸激酶信號轉導以及T細胞活化的負調節劑的作用。[35] Y363的磷酸化可減輕自身抑制並增強與E2的結合。[35] 研究表明,突變會導致Cbl蛋白因失去連接酶/腫瘤抑制功能以及無法維持其正信號傳導/致癌功能而失去功能,從而導致癌症的發展。[36][37]
作為藥物靶標
編輯泛素連接酶底物的篩選
編輯E3-底物相互作用的失調是許多人類疾病的主要原因,因此鑑定E3連接酶底物至關重要。2008年,開發了「全球蛋白質穩定性(GPS)分析」來發現E3泛素連接酶底物。[38]這種高通量系統利用了與數千種潛在底物獨立融合的報告蛋白。通過抑制連接酶活性(通過使Cul1顯性失活,從而使泛素化不發生),報告活性的增加表明已鑑定的底物正在積累。這種方法將大量新底物添加到E3連接酶底物列表中。
可能的治療應用
編輯挑戰
編輯尋找一種選擇性抑制某種E3泛素連接酶活性和/或與疾病有關的蛋白質-蛋白質相互作用的特定分子仍然是重要且不斷擴展的研究領域之一。此外,由於泛素化是一個多步驟過程,涉及各種參與者和中間形式,因此在設計小分子抑制劑時需要充分考慮成分之間非常複雜的相互作用。
類似蛋白質
編輯參閱
編輯參考文獻
編輯- ^ 泛素蛋白. 術語在線. 全國科學技術名詞審定委員會. (簡體中文)
- ^ 泛蛋白. 樂詞網. 國家教育研究院 (中文(臺灣)).
- ^ Wilkinson KD. The discovery of ubiquitin-dependent proteolysis. Proceedings of the National Academy of Sciences of the United States of America. October 2005, 102 (43): 15280–2. Bibcode:2005PNAS..10215280W. PMC 1266097 . PMID 16230621. doi:10.1073/pnas.0504842102 .
- ^ 4.0 4.1 The Nobel Prize in Chemistry 2004. Nobelprize.org. [2010-10-16].
- ^ The Nobel Prize in Chemistry 2004: Popular Information. Nobelprize.org. [2013-12-14].
- ^ Goldstein G, Scheid M, Hammerling U, Schlesinger DH, Niall HD, Boyse EA. Isolation of a polypeptide that has lymphocyte-differentiating properties and is probably represented universally in living cells. Proceedings of the National Academy of Sciences of the United States of America. January 1975, 72 (1): 11–5. Bibcode:1975PNAS...72...11G. PMC 432229 . PMID 1078892. doi:10.1073/pnas.72.1.11 .
- ^ Sharp, PM; Li, WH. Molecular evolution of ubiquitin genes.. Trends in Ecology & Evolution. November 1987, 2 (11): 328–32. PMID 21227875. doi:10.1016/0169-5347(87)90108-X.
- ^ Kimura Y, Tanaka K. Regulatory mechanisms involved in the control of ubiquitin homeostasis. Journal of Biochemistry. June 2010, 147 (6): 793–8. PMID 20418328. doi:10.1093/jb/mvq044 .
- ^ Wilkinson KD. Ubiquitination and deubiquitination: targeting of proteins for degradation by the proteasome. Semin Cell Dev Biol. 2000 Jun;11(3):141-8. doi: 10.1006/scdb.2000.0164. PMID 10906270.
- ^ Popovic, D. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. November 2014, 20 (11): 1242–1253. PMID 25375928. S2CID 205394130. doi:10.1038/nm.3739.
- ^ Yerbury, Justin. Proteome Homeostasis Dysfunction: A Unifying Principle in ALS Pathogenesis. Trends in Neurosciences. May 2020, 43 (5): 274–284. PMID 32353332. S2CID 216095994. doi:10.1016/j.tins.2020.03.002.
- ^ UBQLN1 ubiquilin 1 [ Homo sapiens ]. Gene. National Center for Biotechnology Information. [9 May 2012].
- ^ 13.0 13.1 Stieren ES, El Ayadi A, Xiao Y, Siller E, Landsverk ML, Oberhauser AF, Barral JM, Boehning D. Ubiquilin-1 is a molecular chaperone for the amyloid precursor protein. The Journal of Biological Chemistry. October 2011, 286 (41): 35689–98. PMC 3195644 . PMID 21852239. doi:10.1074/jbc.M111.243147 .
- Alzheimer's brains found to have lower levels of key protein. ScienceDaily (新聞稿). September 1, 2011.
- ^ Heaton SM, Borg NA, Dixit VM. Ubiquitin in the activation and attenuation of innate antiviral immunity. The Journal of Experimental Medicine. January 2016, 213 (1): 1–13. PMC 4710203 . PMID 26712804. doi:10.1084/jem.20151531.
- ^ Takeuchi O, Akira S. Pattern recognition receptors and inflammation. Cell. March 2010, 140 (6): 805–20. PMID 20303872. S2CID 223338. doi:10.1016/j.cell.2010.01.022 (英語).
- ^ Okamoto M, Kouwaki T, Fukushima Y, Oshiumi H. Regulation of RIG-I Activation by K63-Linked Polyubiquitination. Frontiers in Immunology. 2018, 8: 1942. PMC 5760545 . PMID 29354136. doi:10.3389/fimmu.2017.01942 (英語).
- ^ Huber C, Dias-Santagata D, Glaser A, O'Sullivan J, Brauner R, Wu K, Xu X, Pearce K, Wang R, Uzielli ML, Dagoneau N, Chemaitilly W, Superti-Furga A, Dos Santos H, Mégarbané A, Morin G, Gillessen-Kaesbach G, Hennekam R, Van der Burgt I, Black GC, Clayton PE, Read A, Le Merrer M, Scambler PJ, Munnich A, Pan ZQ, Winter R, Cormier-Daire V. Identification of mutations in CUL7 in 3-M syndrome. Nature Genetics. October 2005, 37 (10): 1119–24. PMID 16142236. S2CID 44003147. doi:10.1038/ng1628.
- ^ Nguyen LK, Kolch W, Kholodenko BN. When ubiquitination meets phosphorylation: a systems biology perspective of EGFR/MAPK signalling. Cell Communication and Signaling. July 2013, 11: 52. PMC 3734146 . PMID 23902637. doi:10.1186/1478-811X-11-52 .
- ^ Sorkin A, Goh LK. Endocytosis and intracellular trafficking of ErbBs. Experimental Cell Research. October 2008, 314 (17): 3093–106. PMC 2605728 . PMID 18793634. doi:10.1016/j.yexcr.2008.07.029.
- ^ Nguyen LK, Muñoz-García J, Maccario H, Ciechanover A, Kolch W, Kholodenko BN. Switches, excitable responses and oscillations in the Ring1B/Bmi1 ubiquitination system. PLOS Computational Biology. December 2011, 7 (12): e1002317. Bibcode:2011PLSCB...7E2317N. PMC 3240587 . PMID 22194680. doi:10.1371/journal.pcbi.1002317 .
- ^ Zhou W, Wang X, Rosenfeld MG. Histone H2A ubiquitination in transcriptional regulation and DNA damage repair. The International Journal of Biochemistry & Cell Biology. January 2009, 41 (1): 12–5. PMID 18929679. doi:10.1016/j.biocel.2008.09.016.
- ^ Dou QP, Li B. Proteasome inhibitors as potential novel anticancer agents. Drug Resistance Updates. August 1999, 2 (4): 215–223. PMID 11504494. doi:10.1054/drup.1999.0095 .
- ^ Vries EG, Verweij J. Clinical Cancer Research 2000: New Agents and Therapies. Drug Resistance Updates. 2000, 3 (4): 197–201. PMID 11498385. doi:10.1054/drup.2000.0153.
- ^ Pray TR, Parlati F, Huang J, Wong BR, Payan DG, Bennett MK, Issakani SD, Molineaux S, Demo SD. Cell cycle regulatory E3 ubiquitin ligases as anticancer targets. Drug Resistance Updates. December 2002, 5 (6): 249–58. PMID 12531181. doi:10.1016/s1368-7646(02)00121-8.
- ^ Clifford SC, Cockman ME, Smallwood AC, Mole DR, Woodward ER, Maxwell PH, Ratcliffe PJ, Maher ER. Contrasting effects on HIF-1alpha regulation by disease-causing pVHL mutations correlate with patterns of tumourigenesis in von Hippel–Lindau disease. Human Molecular Genetics. 2001, 10 (10): 1029–38. PMID 11331613. doi:10.1093/hmg/10.10.1029 .
- ^ Sparks AB, Morin PJ, Vogelstein B, Kinzler KW. Mutational analysis of the APC/beta-catenin/Tcf pathway in colorectal cancer. Cancer Research. March 1998, 58 (6): 1130–4. PMID 9515795.
- ^ Scheffner M, Huibregtse JM, Vierstra RD, Howley PM. The HPV-16 E6 and E6-AP complex functions as a ubiquitin-protein ligase in the ubiquitination of p53. Cell. November 1993, 75 (3): 495–505. PMID 8221889. S2CID 27437768. doi:10.1016/0092-8674(93)90384-3.
- ^ Momand J, Jung D, Wilczynski S, Niland J. The MDM2 gene amplification database. Nucleic Acids Research. August 1998, 26 (15): 3453–9. PMC 147746 . PMID 9671804. doi:10.1093/nar/26.15.3453.
- ^ Hashizume R, Fukuda M, Maeda I, Nishikawa H, Oyake D, Yabuki Y, Ogata H, Ohta T. The RING heterodimer BRCA1-BARD1 is a ubiquitin ligase inactivated by a breast cancer-derived mutation. The Journal of Biological Chemistry. May 2001, 276 (18): 14537–40. PMID 11278247. doi:10.1074/jbc.C000881200 .
- ^ Zhu CQ, Blackhall FH, Pintilie M, Iyengar P, Liu N, Ho J, Chomiak T, Lau D, Winton T, Shepherd FA, Tsao MS. Skp2 gene copy number aberrations are common in non-small cell lung carcinoma, and its overexpression in tumors with ras mutation is a poor prognostic marker. Clinical Cancer Research. 2004, 10 (6): 1984–91. PMID 15041716. doi:10.1158/1078-0432.ccr-03-0470 .
- ^ Schmidt MH, Furnari FB, Cavenee WK, Bögler O. Epidermal growth factor receptor signaling intensity determines intracellular protein interactions, ubiquitination, and internalization. Proceedings of the National Academy of Sciences of the United States of America. May 2003, 100 (11): 6505–10. Bibcode:2003PNAS..100.6505S. PMC 164476 . PMID 12734385. doi:10.1073/pnas.1031790100 .
- ^ 32.0 32.1 Knuutila S, Aalto Y, Autio K, Björkqvist AM, El-Rifai W, Hemmer S, Huhta T, Kettunen E, Kiuru-Kuhlefelt S, Larramendy ML, Lushnikova T, Monni O, Pere H, Tapper J, Tarkkanen M, Varis A, Wasenius VM, Wolf M, Zhu Y. DNA copy number losses in human neoplasms. The American Journal of Pathology. September 1999, 155 (3): 683–94. PMC 1866903 . PMID 10487825. doi:10.1016/S0002-9440(10)65166-8.
- ^ Mani A, Gelmann EP. The ubiquitin-proteasome pathway and its role in cancer. Journal of Clinical Oncology. July 2005, 23 (21): 4776–89. PMID 16034054. doi:10.1200/JCO.2005.05.081.
- ^ Witowsky JA, Johnson GL. Ubiquitylation of MEKK1 inhibits its phosphorylation of MKK1 and MKK4 and activation of the ERK1/2 and JNK pathways. The Journal of Biological Chemistry. January 2003, 278 (3): 1403–6. PMID 12456688. doi:10.1074/jbc.C200616200 .
- ^ 35.0 35.1 35.2 Kobashigawa Y, Tomitaka A, Kumeta H, Noda NN, Yamaguchi M, Inagaki F. Autoinhibition and phosphorylation-induced activation mechanisms of human cancer and autoimmune disease-related E3 protein Cbl-b. Proceedings of the National Academy of Sciences of the United States of America. December 2011, 108 (51): 20579–84. Bibcode:2011PNAS..10820579K. PMC 3251137 . PMID 22158902. doi:10.1073/pnas.1110712108 .
- ^ Niemeyer CM, Kang MW, Shin DH, Furlan I, Erlacher M, Bunin NJ, Bunda S, Finklestein JZ, Sakamoto KM, Gorr TA, Mehta P, Schmid I, Kropshofer G, Corbacioglu S, Lang PJ, Klein C, Schlegel PG, Heinzmann A, Schneider M, Starý J, van den Heuvel-Eibrink MM, Hasle H, Locatelli F, Sakai D, Archambeault S, Chen L, Russell RC, Sybingco SS, Ohh M, Braun BS, Flotho C, Loh ML. Germline CBL mutations cause developmental abnormalities and predispose to juvenile myelomonocytic leukemia. Nature Genetics. September 2010, 42 (9): 794–800. PMC 4297285 . PMID 20694012. doi:10.1038/ng.641.
- ^ Kales SC, Ryan PE, Nau MM, Lipkowitz S. Cbl and human myeloid neoplasms: the Cbl oncogene comes of age. Cancer Research. June 2010, 70 (12): 4789–94. PMC 2888780 . PMID 20501843. doi:10.1158/0008-5472.CAN-10-0610.
- ^ Yen HC, Elledge SJ. Identification of SCF ubiquitin ligase substrates by global protein stability profiling. Science. 2008, 322 (5903): 923–9. Bibcode:2008Sci...322..923Y. PMID 18988848. S2CID 23586705. doi:10.1126/science.1160462.
外部連結
編輯- GeneReviews/NCBI/NIH/UW entry on Angelman syndrome
- OMIM entries on Angelman syndrome
- UniProt entry for ubiquitin
- 7.340 Ubiquitination: The Proteasome and Human Disease. MIT OpenCourseWare. 2004. Notes from MIT course.
- 醫學主題詞表(MeSH):Ubiquitin