古菌
古菌(拉丁语:Archaea,来自古希腊语:ἀρχαῖα,意为“古代的东西”)又称古核生物[4],旧称古细菌[5](Archaebacteria)、古生菌、太古生物,是单细胞微生物,构成生物分类的一个域,或一个界。这些微生物1970年前的分类属于原核生物,它们与细菌有很多相似之处,即它们没有细胞核与任何其他膜状胞器,同时另一些特征相似于真核生物,比如存在重复序列与核小体。
| 古菌域 化石时期:太古代-现今
| |
|---|---|
| |
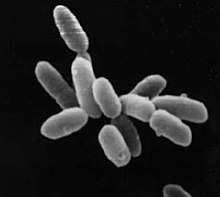
| NRC-1系盐杆菌纲,每一细胞长度大约5μm | |
| 科学分类 | |
| 域: | 古菌域 Archaea Woese, Kandler & Wheelis, 1990[1] |
| 界[2]与门[3] | |
过去曾经将古菌和细菌一同归为原核生物,并将其命名为“古细菌”,但这种分类方式已过时[6]。事实上古菌有其独特的进化历程,并与其它生命形式有显著的生化差异,所以现在将其列为三域系统中的一个域。在这个系统中,古菌、细菌与真核生物各为一个域,并进一步划分为界与门。到目前为止,古菌已被划分为公认的四个门,随着进一步研究,还可能建立更多的门类。在这些类群中,研究最深入的是泉古菌门与广古菌门。但对古菌进行分类仍然是困难的,因为绝大多数的古菌都无法在实验室中纯化培养,只能通过环境宏基因组检测来分析。
古菌和细菌的大小和形状非常相似,但少数古菌有不寻常的形状,如嗜盐古菌窝氏盐方扁平古菌拥有平面正方形的细胞。[7][8]尽管看起来与细菌更相似,但古菌与真核生物的亲缘关系更为密切,特别是在一些代谢途径(如转录和转译)有关酶的相似性上。古菌还有一些性状是独一无二的,比如由依赖醚键构成的细胞膜。与真核生物相比,古菌有更多的能量来源,从熟悉的有机物糖类到氨到金属离子直到氢气。嗜盐菌,如嗜卤盐菌可以以太阳光为能源,其它一些种类的古菌能进行固碳反应;没有一种古菌能像蓝藻与植物进行光合作用。古菌通过分裂、出芽、断裂来进行无性生殖,但没有发现能产生孢子的种类。
一开始,古菌被认为都是一些生活在温泉、盐湖之类极端环境的嗜极生物,但近来发现它们的栖息地其实十分广泛,从土壤、海洋、到河流湿地。它们也被发现在人类的大肠、口腔、与皮肤[9]。尤其是在海洋中古菌特别多,一些浮游生物中的古菌可能是这个星球上数量最大的生物群体。现在,古菌被认为是地球生命的一个重要组成部分,在碳循环和氮循环中可能扮演重要的角色。目前没有已知的作为病原体或寄生虫的古菌,他们往往是偏利共生或互利共生。一个例子是产甲烷菌,生活在人和反刍动物的肠道中帮助消化,产甲烷菌还被用于沼气生产和污水处理。嗜极生物古菌中的酶能承受高温和有机溶剂,常被生物技术所利用。
历史
编辑新域的诞生
编辑在20世纪的大部分时间里,原核生物因其生化、形态和代谢上的一致性被视为分类上的单组生物。在这一时期,微生物学家试图根据其形状、细胞壁结构以及所消耗的物质来对微生物进行分类[10]。然而,在1965年有一种新方法被提出[11],即使用生物体的基因序列来搞清这些生物是如何相关联的。这种方法称为系统发育,是今天生物分类所使用的主要方法。
“古细菌”这个概念第一次出现是在1977年,由卡尔·乌斯和乔治·福克斯提出的,原因是它们在16S 核糖体RNA的系统发生树上和其它原核生物的区别[12]。这两组原核生物起初被定为古细菌和真细菌两个界或亚界(当时乌斯与福克斯更倾向于用“总界”一词)。后来乌斯认为它们是两支根本不同的生物,于是在1990年重新命名其为古菌和细菌[1],并将这两支和真核生物划为域这一新建立的分类阶元,一起构成了生物的三域系统。
起初只有一些产甲烷菌属于这个新域,这些古菌以一些极端环境为栖息地,比如温泉和盐湖。到20世纪末,微生物学家意识到古菌是一个庞大而多元化的群体,不只局限于极端环境,而是广泛存在于自然界中,如土壤和海洋里[13]。由于聚合酶链式反应(PCR)被大量用于探测采样土壤或水中的原核生物的核酸,古菌的重要性和独特性得到新的重视。PCR方法可以不经细菌培养而直接探测和鉴别微生物[14][15]。
现行分类
编辑古菌和原核生物的分类一般是一个快速发展并充满争议的领域。现行的分类体系旨在将古菌组织成具有共同结构特征和共同祖先的生物群[16]。这些分类很大程度上依赖于使用核糖体RNA基因序列来揭示生物之间的关系(分子系统发生学)[17]。目前大部分可纯化培养的并研究清楚的古菌都可以划为两个主要的门:泉古菌门和广古菌门。其它的门类还在初步创立阶段,比如2002年由卡尔·施泰特尔发现的奇特的物种骑行纳古菌而建立的纳古菌门[18]。还有1994年Barns等建议建立的一个新门初古菌门包括一小群不寻常的嗜热品种,但这个门与泉古菌门演化关系密切[19][20]。其它最近探别出来的古菌与已知古菌门类关系较远,如2006年发现的环境样品ARMAN[21][22]。ARMAN是已知的生命体中最小的生命体[22]。
一个超门(superphylum)分类-TACK(又称变形古菌界),已经被提出,包括奇古菌门,曙古菌门,泉古菌门和初古菌门[23]。这个超门可能与真核生物的起源有关。最近,阿斯加德古菌超门被命名,并被提议与真核生物的起源和一个TACK姊妹组织更紧密相关[24]。
细菌、古菌和真核生物的比较
编辑在细胞结构和代谢上,古菌在很多方面接近其它原核生物。然而在基因转录和转译这两个分子生物学的中心过程上,它们并不明显表现出细菌的特征,反而非常接近真核生物。比如,古菌的转译使用真核生物的启动和延伸因子,且转译过程需要真核生物中的TATA框结合蛋白和TFIIB。许多这些特征也被讨论如下。
古菌还具有一些其它特征。与大多数细菌不同,它们只有一层细胞膜而缺少肽聚糖细胞壁。而且,绝大多数细菌和真核生物的细胞膜中的脂类主要由甘油酯组成,而古菌的膜脂由甘油醚构成。这些区别也许是对超高温环境的适应。古菌鞭毛的成分和形成过程也与细菌不同。
| 与细菌相同 | 与真核生物相同 | 古菌独有 |
|---|---|---|
| 没有细胞核和膜结合细胞器 | 没有肽聚糖 | 独特的细胞壁结构 |
| 环状基因组 | DNA与组蛋白结合[25][26] | 细胞膜由醚键构成 |
| 基因组成操纵子 | 翻译从甲硫氨酸起始 | 鞭毛蛋白结构[27] |
| 没有像真核生物一样复杂的转录后修饰 | 相似的RNA聚合酶、启动子以及其它转录机制[27][28][29] | 核糖体结构 |
| 多顺反子mRNA | 相似的DNA复制与修复[30] | tRNA的序列和代谢[27][31] |
| 细胞大小(远小于真核生物) | 相似的ATP酶 | 独有的紫膜光合系统 |
古菌和真核生物的关系仍然是个重要问题。除掉上面所提到的相似性,很多其他遗传树也将二者并在一起。在一些树中真核生物离广古菌比离泉古菌更近,但生物膜化学的结论相反。然而,在一些细菌,(如栖热袍菌)中发现了和古菌类似的基因,使这些关系变得复杂起来。一些人认为真核生物起源于一个古菌和细菌的融合,二者分别成为细胞核和细胞质。这解释了很多基因上的相似性,但在解释细胞结构上存在困难。
形态
编辑单个古菌细胞直径在0.1到15微米之间,有一些种类形成细胞团簇或者纤维,长度可达200微米。它们可有各种形状,一般是球形、杆形、螺旋形、叶状或方形。这些不寻常的形状可能是由它们的细胞壁和原核生物细胞骨架两者维持的。其他生物的细胞骨架成分蛋白在古菌内存在[32],长丝在其细胞内形成[33],但与其他生物体不同的是,这些细胞结构被理解的很少[34]。
细胞结构、组成和运行
编辑古菌和细菌的细胞结构大体类似。古菌和细菌一样没有内膜系统和细胞器[35]。古菌也和细菌一样在细胞膜之外还有一层细胞壁,它们同样也具有一个或多个鞭毛结构[36]。
膜
编辑古菌膜由从那些在其它生命形式的强烈不同的分子组成,显示出古菌与细菌和真核生物关系都比较遥远。
细胞壁和鞭毛
编辑大多数古菌(但不是Thermoplasma属和Ferroplasma属)具有一个细胞壁。[37]在大多数古细菌的壁由表面层蛋白质组成,形成一个S层。一个S层是蛋白质分子的刚性阵列覆盖了细胞的外侧(如同锁子甲)。[38]该层提供化学和物理保护,并能防止大分子接触细胞膜。[39]与细菌不同,古菌的细胞壁缺少肽聚糖。[40]
古菌鞭毛操作类似细菌鞭毛,它们的长梗是由在座部的旋转马达驱动。然而,古菌鞭毛在组成和发育上有着明显的不同。[36]这两种类型的鞭毛是从不同的祖先进化而来的。细菌鞭毛与III型分泌系统共享一个共同的祖先,[41][42]但是,古菌鞭毛似乎已经从细菌IV型菌的性菌毛进化而来。[43]对比,细菌鞭毛是空心的,并且是由其子单元从中心孔向上移动到鞭毛的末端装配,但是,古菌鞭毛是由在基座中添加子单元合成。[44]
遗传学
编辑古菌通常拥有单个环状的染色体[45],其大小差异悬殊。目前已知最大的是Methanosarcina acetivorans的基因组,达5,751,492个碱基对[46],而除里奇蒙德矿井嗜酸纳米古生物(ARMAN)外最小的骑行纳古菌只有490,885个碱基对,不到前者的十分之一,据估只能编码537种蛋白质[47]。一些小型的独立DNA片段质粒也存在于古菌中,可能通过细胞之间的直接接触传递,其过程可能与细菌之间的接合类似[48][49]。
古菌可被双链DNA病毒感染,这些双链DNA病毒形态各异,包括瓶状、杆状、雨滴状等[51]。这些病毒在一些嗜热菌中研究得最为详致,特别是硫化叶菌目和热变形菌目的古菌中[52]。最近发现了两类可感染古菌的单链DNA病毒:一种是典型的嗜盐菌多形病毒,这病毒感染嗜盐菌[53]另外一种嗜热的圈形病毒感染嗜超热菌(这些古菌生长在90到95°C的温度下)[54]其中后者有已知最大的单链DNA基因组。古菌可以利用针对病毒基因的RNA干扰和DNA重复影响病毒在寄主内的繁殖。[55][56]
古菌在基因上与细菌真核生物有许多不同,大约有15%古菌基因是古菌中独有的。这些古菌独有蛋白质的功能大部分还未知[57],其余已知功能的独有蛋白质大部分属于广古菌门中的产甲烷菌。古菌、细菌、真核生物共有的蛋白质都涉及细胞的核心功能,如转录、翻译和核苷酸代谢。古菌基因组别的特点还有基因的组织常与功能相关(如催化相同代谢途径的酶会组成一个独有的操纵子),以及不同的tRNA基因之间差异很大,其氨酰-tRNA合成酶之间也是如此[58]。
较之细菌的转录与翻译,古菌中的这些过程更接近于真核生物,特别是古菌中的RNA聚合酶与核糖体,可以说非常接近于真核生物中的对应物[45]。尽管古菌中只有一种RNA聚合酶,但其结构与功能与真核生物中最主要的RNA聚合酶Ⅱ非常相似,且其结合到基因启动子上所需的蛋白质组件(通用转录因子)也非常相似[59]。但是,其它的古菌转录因子却与细菌中的相似[60],转录后修饰也比真核生物中的简单(大部分古菌基因缺少内含子,其内含子主要集中在tRNA和rRNA基因中[61],少数出现在编码蛋白质的基因中[62][63])。
目前已有一百五十余个古菌基因组已经完成了测序,另外许多测序工作正在进行中。[64]
基因的转移与交换
编辑沃氏富盐菌一种极端嗜盐古菌,可在两个细胞的细胞质之间形成桥梁,可能用于DNA的任意方向的转移[65]。
当嗜热的古菌硫化叶菌[66]和嗜酸热硫化叶菌[67]在DNA突变源,例如紫外线、博来霉素和丝裂霉素的暴露下,不同种类的古菌会出现不同的变化。硫化叶菌的基因变化不会因为其他物理突变源,如酸碱值和温度,[66]这说明了古菌的进化是来自于DNA的损伤,[67]这也说明了紫外线导致的细胞进化促使染色体在硫化叶菌之间交换。重组的几率级别比没有经过紫外线照射的古菌多出三级。[66][67][68]这使科学家猜测可能硫化叶菌为了维修它们自己损坏的DNA而与有类似基因的硫化叶菌交换质粒。这反应可能是原始的性交流,这与被经常研究的细菌固定的DNA交换以维修受损DNA类似。[69]
繁殖
编辑古菌利用二分裂、分裂和出芽进行无性繁殖;古菌不会进行减数分裂,所以拥有同样的基因的一个种的古菌可能拥有不同的形态[70]。古菌的细胞分裂被它们的细胞周期所控制;在细胞的染色体复制并分离后,细胞开始一分为二[71]。虽说目前古菌中只有硫化叶菌的复制周期被阐明,但是这样的细胞周期大体上类似于在细菌和真核生物的周期。和真核生物一样,古菌的染色体也可以在多个位点(复制起点)开始用DNA聚合酶复制[72]。然而古菌用于控制细胞分裂的蛋白(例如FtsZ,制造逐渐缩小的“Z环”以帮助细胞质分裂)和分离两个子细胞的隔膜部分,这些与细菌的二分裂相似[71]。
但与细菌、真核生物不同,目前未发现有古菌进行孢子生殖[73]。一些盐杆菌纲的种类可以进行表型转换并生长成为不同的形态。这些形态包括拥有可以防止渗透压休克出现的厚细胞壁,这使嗜卤盐菌可以在盐度低的水中存活。这些古菌特征不是生殖结构,但是它们可以帮助古菌在新的环境下生存[74]。
生态学
编辑生活环境
编辑古菌可以在各种各样的栖息地中出现并且是全球生态重要的一部分[13],古菌占有地球上20%的生物量。[75]很多古菌是生存在极端环境中的,包括最早发现的古菌也是嗜极生物。[76]一些生存在极高的温度(经常100 °C以上)下,比如间歇泉、石油井或者海底深海热泉中。还有的生存在很冷的环境或者高盐、强酸或强碱性的水中。然而也有些古菌是嗜中性的,能够在沼泽、废水、海洋和土壤中被发现。[13]很多产甲烷的古菌生存在动物的消化道中,如反刍动物、白蚁或者人类。古菌通常对其它生物无害,且未知有致病古菌。
极端环境下的古菌主要可以分为四种生理群:嗜盐生物、嗜热生物、嗜碱生物及嗜酸生物[77]。这些不是具体的分类,这些分类也不是互斥的,因此有些古菌有时属于其中的几类中,不过这仍然可以做为分类的起点。
嗜盐类的古菌,生活在高含盐量的环境中,例如盐湖,盐度比嗜盐细菌可以生活的20-25%要高[76]。嗜热古菌适合生长在超过45 °C(113 °F)的温度,例如热泉中,超嗜热古菌在温度超过80 °C(176 °F)时生长的最好[78]。古菌Methanopyrus kandleri的116菌种甚至122 °C(252 °F)繁殖,是生物中最高的记录[79]。
有些古菌可以生长在极酸性或极碱性的环境中[77],例如嗜酸古菌中的Picrophilus torridus可以生长在pH值为0的环境下,相当于1.2 M硫酸[80]。
古菌可以抵抗极端环境的能力也让科学家推测外星生命具有的可能的特质[81],有些极端的环境和火星的环境有些相近。[82],因此推测这些古菌有可能在陨石上,在各行星之间移动[83]。
近来许多研究发现古菌不只可在高温及中温的环境下生存,也可以在极低温的环境下生存,例如在极地的海洋中就有许多的古菌[84],不过数量更多的是在海洋非极端环境中的古菌,是浮游生物中的微微型浮游生物[85]。虽然这种古菌数量很多(占纯生物生物量的40%),但其中几乎都无法在实验室隔离的进行纯培养[86]。因此古菌在海洋生态中的角色是基础的,古菌对整体生物地球化学循环的影响大部分都还不清楚。[87]有些海洋的奇古菌可以产生硝化作用,因此推测对海洋的氮循环应该有影响[88],不过这些泉古菌也会使用其他的能量来源[89]。在海床上的沉积物中可以找到大量的古菌,这也是在海底深度超过一米的区域中,在数量上占大多数的生物体[90]。
在地球的化学循环中的作用
编辑古菌参与了地球上碳、氮、硫的循环。虽说这些细胞活动对生态系统来说是十分重要的,但是古菌也可能制造与人为变化类似的环境影响,甚至可能造成环境污染。
古菌也可以进行大部分氮循环中的化学反应。有一些古菌可以从一个生态系统中移除有机氮,这些化学反应包括一些硝酸盐代谢和反硝化反应;其它古菌可以把氮气加入生态形成有机氮,这些反应包括氮吸收和固氮作用。[91][92]最近,科学家发现古菌也参与氨的氧化。这些反应在海洋里是特别重要的。[93][94]一些古菌也参与土壤里面的氨氧化。这些古菌会制造硝酸盐,而其它微生物则进一步氧化硝酸盐。最后,植物和其它生物会吸收并利用氧化的硝酸盐。[95]
在硫循环中,利用硫化物氧化代谢的古菌可以把硫从岩石中释放出来,并提供给其它生物。但是这些古菌,例如硫化叶菌,会利用硫代谢并最终产出硫酸,所以这种古菌在废弃的矿场中的生长会导致矿渣酸性废水的出现以及其它环境污染。[96]
在碳循环中,制造甲烷的古菌可以移除多余的氢并且在有机化合物的缺氧分解中扮演着分解者的角色。这些古菌可以在沉积物和沼泽等缺氧环境中生活,它们也可以被用于污水处理[97]
产甲烷菌是大气甲烷的主要来源,它们每年释放的甲烷占了世界上大部分的甲烷排放。[98]产甲烷菌排放的甲烷加速了全球的温室气体排放和全球变暖。
全球甲烷数值于前工业化时期的722PPB(10亿吨大气内有722吨甲烷)增加到2011年的1800PPB,这期间甲烷数值上升到了之前的2.5倍,2011年是在80万年内全球甲烷数值最高的一年。[99]甲烷的全球变暖潜能值是29,这说明了甲烷可以在100年内吸收比二氧化碳多29倍的热量。[100]
与其它生物的关系
编辑古菌与其它生物之间的关系主要都是互利共生或者偏利共生。至今还没有发现古菌是任何其它生物的病原体或寄生虫。[101][102]虽说至今没有发现并确定古菌病原体,但是研究证明一些产甲烷菌的种类可能与牙周疾病有关。[103][104]另外,一种骑行纳古菌Nanoarchaeum equitans可能是泉古菌适宜火球古菌的寄生物,必须在宿主细胞内生活并繁殖,[105]但是对寄主没有任何好处。[106]相反的,里奇蒙德矿井嗜酸纳米古生物(ARMAN)只是与酸性矿井内生物薄膜的其它古菌偶尔接触。[107]这种关系的性质至今仍然是未知的。但是科学家知道纳古菌门和燃球菌属之间的关系与微小的ARMAN古菌不同,ARMAN古菌一般与嗜酸热菌细胞不会相互影响。
互利共生
编辑所有反刍动物和白蚁的消化道中有其中一种在产甲烷菌和原生动物之间的互利共生,它们帮助寄主消化纤维素。[108]在这缺氧的环境中,原生动物利用纤维素获得能量并同时释放产物氢气。大量的氢气会影响原生动物获得能量的效率。当产甲烷菌把氢气经过化学反应变成甲烷后,原生动物会因为能获得更多能量而获益。[109]
一些古菌生活在厌氧原生动物里面并利用寄主造氢体制造的氢气,这些原生动物包括纤毛虫Plagiopyla frontata。[110][111]古菌也与更大的生物有共生关系。例如海里的古菌Cenarchaeum symbiosum生活在海绵Axinella mexicana的里并与海绵拥有共生关系。[112]
偏利共生
编辑古菌也可以是偏利共生中的获益者,同时寄主既没有获益也没有受害。例如史密斯甲烷菌就是在的人体正常肠道菌群中最常见的古菌,这种古菌占了肠道中原核生物的十分之一[113]。在白蚁和人体内,这些产甲烷菌甚至可能是有益的,这些古菌会与其它菌群互相影响并协助消化[114]。古菌的菌落也与不少其它生物表面或附近生活,例如在珊瑚的表面[115]和环绕植物根冠的根际。[116][117]
在技术与工业中的作用
编辑一些耐热或耐酸碱的嗜极古菌可以作为一些在极端环境下活跃的酶的来源。[118][119]这些来自古菌的酶拥有很多用途。其中耐高温的DNA聚合酶,例如从焦酚火球菌中提取的火球菌DNA聚合酶可以在需要进行聚合酶链式反应时简单并快速地复制DNA,这彻底改变了对分子生物学地研究。在工业中,来自其它焦酚菌的淀粉酶、半乳糖苷酶和普鲁兰酶可以在高达100 °C(212 °F)的环境下仍然活跃,这使食品加工可以在高温下进行,这些酶可以被用来加工低乳糖牛奶和乳清。[120]嗜热古菌生产的酶可以在有机溶剂内仍然保持稳定,这些酶可以在保持环保的前提下用来合成有机化合物。[119]这种稳定性使它们更容易在结构生物学中研究。这就是为什么与细菌和真核生物对应的嗜极古菌生产的酶经常被用于分子结构的研究。[121]
相较于酵素领域的应用,古菌本身在生物科技上的应用就比较少了。产甲烷的古菌是进行厌氧消化,产生生物气体的微生物群体的一部分,是污水处理中重要的一环[122]。在矿石处理中,嗜酸古菌可以用来从矿物中萃取包括金、钴及铜等金属[123]。
古菌也是一些潜在有效抗细菌药的来源,已经分析了其中几种的古菌素,一般认为有几百种,特别是由嗜盐细菌及硫化叶菌所产生的。这些古菌素的结构和从细菌产生的抗细菌药不同,所以可能有新的作用方式。此外它们可以创建新的选择标记,可以用在古菌分子生物学中[124]。
新分类
编辑“Miscellaneous Crenarchaeota Group” (MCG)古菌是迄今为止发现分布最为广泛的一类未培养古菌,不仅分布广泛,而且数量庞大,被认为是海底深部生物圈中最丰富,并且最活跃的类群之一,很可能在全球物质和能量循环过程中发挥了重要的作用。然而到目前为止,尚未获得此类古菌的纯培养,其生理和功能特征以及在环境中发挥的作用都不得而知,并且其分类地位也不明确,暂时归类于泉古菌门。研究者们发现MCG古菌在深部生物圈沉积物中广泛并大量存在,代表了一类自然界较古老的古菌,在系统发育上处于一个深的分支,显著不同于目前分类已确定的所有古菌门类,包括新近确立的奇古菌门和曙古菌门。因此,提议将MCG古菌归类于一个全新的门类,命名为深古菌门(Bathyarchaeota)。是古菌和生命起源和演化研究的重要进展之一,也将为本领域的科学研究起到积极推动作用。
本域包括以下门:[125]
- Methanobacteriota Garrity and Holt 2023
- Microcaldota Sakai et al. 2023
- Nanobdellota Huber et al. 2023
- Nanobdellia Kato et al. 2022
- Nanobdellales Kato et al. 2022
- Nanobdellaceae Kato et al. 2022
- Nanobdella Kato et al. 2022
- Nanobdella aerobiophila Kato et al. 2022[126]
- Nanobdella Kato et al. 2022
- Nanobdellaceae Kato et al. 2022
- Nanobdellales Kato et al. 2022
- Nanobdellia Kato et al. 2022
- Thermoproteota Garrity and Holt 2021
参见
编辑参考文献
编辑- ^ 1.0 1.1 Woese, C R; Kandler, O; Wheelis, M L. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya.. Proceedings of the National Academy of Sciences. 1990-06, 87 (12) [2022-10-13]. Bibcode:1990PNAS...87.4576W. ISSN 0027-8424. PMC 54159 . PMID 2112744. doi:10.1073/pnas.87.12.4576. (原始内容存档于2023-09-16) (英语).
- ^ Petitjean, Céline; Deschamps, Philippe; López-García, Purificación; Moreira, David. Rooting the Domain Archaea by Phylogenomic Analysis Supports the Foundation of the New Kingdom Proteoarchaeota. Genome Biology and Evolution. 2015-01, 7 (1) [2022-10-13]. ISSN 1759-6653. PMC 4316627 . PMID 25527841. doi:10.1093/gbe/evu274. (原始内容存档于2022-10-13) (英语).
- ^ NCBI taxonomy page on Archaea. [2017-04-27]. (原始内容存档于2020-12-01).
- ^ 存档副本. [2023-07-24]. (原始内容存档于2023-07-24).
- ^ 存档副本. [2023-07-24]. (原始内容存档于2023-07-24).
- ^ Pace, Norman R. Time for a change. Nature. 2006-05, 441 (7091) [2022-10-13]. Bibcode:2006Natur.441..289P. ISSN 0028-0836. PMID 16710401. doi:10.1038/441289a. (原始内容存档于2019-04-11) (英语).
- ^ 鹽方扁平古菌:生活在鹽田裡的天然小方塊光電板. PanSci 泛科学. [2022-10-13]. (原始内容存档于2022-10-17) (中文(台湾)).
- ^ Stoeckenius, W. Walsby's square bacterium: fine structure of an orthogonal procaryote. Journal of Bacteriology. 1981-10, 148 (1) [2022-10-13]. ISSN 0021-9193. PMC 216199 . PMID 7287626. doi:10.1128/jb.148.1.352-360.1981. (原始内容存档于2022-10-17) (英语).
- ^ Bang, Corinna; Schmitz, Ruth A. Narberhaus, Franz , 编. Archaea associated with human surfaces: not to be underestimated. FEMS Microbiology Reviews. 2015-09, 39 (5) [2022-10-13]. ISSN 1574-6976. PMID 2590711. doi:10.1093/femsre/fuv010. (原始内容存档于2023-01-23) (英语).
- ^ Staley, James T. The bacterial species dilemma and the genomic–phylogenetic species concept. Philosophical Transactions of the Royal Society B: Biological Sciences. 2006-11-29, 361 (1475) [2022-10-13]. ISSN 0962-8436. PMC 1857736 . PMID 17062409. doi:10.1098/rstb.2006.1914. (原始内容存档于2022-09-30) (英语).
- ^ Zuckerkandl, Emile; Pauling, Linus. Molecules as documents of evolutionary history. Journal of Theoretical Biology. 1965-03-01, 8 (2) [2022-10-13]. ISSN 0022-5193. PMID 5876245. doi:10.1016/0022-5193(65)90083-4. (原始内容存档于2018-12-08) (英语).
- ^ Woese, Carl R.; Fox, George E. Phylogenetic structure of the prokaryotic domain: The primary kingdoms. Proceedings of the National Academy of Sciences. 1977-11, 74 (11) [2022-10-13]. Bibcode:1977PNAS...74.5088W. ISSN 0027-8424. doi:10.1073/pnas.74.11.5088. (原始内容存档于2022-10-13) (英语).
- ^ 13.0 13.1 13.2 DeLong, Edward F. Everything in moderation: Archaea as ‘non-extremophiles’. Current Opinion in Genetics & Development. 1998-12-01, 8 (6). ISSN 0959-437X. PMID 9914204. doi:10.1016/S0959-437X(98)80032-4 (英语).
- ^ Theron, J.; Cloete, T. E. Molecular Techniques for Determining Microbial Diversity and Community Structure in Natural Environments. Critical Reviews in Microbiology. 2000-01, 26 (1). ISSN 1040-841X. PMID 10782339. doi:10.1080/10408410091154174 (英语).
- ^ Schmidt, Thomas M. The maturing of microbial ecology. International Microbiology: The Official Journal of the Spanish Society for Microbiology. 2006-09, 9 (3) [2022-10-13]. ISSN 1139-6709. PMID 17061212. (原始内容存档于2022-10-14).
- ^ Gevers, Dirk; Dawyndt, Peter; Vandamme, Peter; Willems, Anne; Vancanneyt, Marc; Swings, Jean; De Vos, Paul. Stepping stones towards a new prokaryotic taxonomy. Philosophical Transactions of the Royal Society B: Biological Sciences. 2006-11-29, 361 (1475) [2022-10-13]. ISSN 0962-8436. PMC 1764938 . PMID 17062410. doi:10.1098/rstb.2006.1915. (原始内容存档于2022-10-17).
- ^ Robertson, Charles E; Harris, J Kirk; Spear, John R; Pace, Norman R. Phylogenetic diversity and ecology of environmental Archaea. Current Opinion in Microbiology. Growth development / edited by John N Reeve and Ruth A Schmitz. 2005-12-01, 8 (6). ISSN 1369-5274. PMID 16236543. doi:10.1016/j.mib.2005.10.003 (英语).
- ^ Huber, Harald; Hohn, Michael J.; Rachel, Reinhard; Fuchs, Tanja; Wimmer, Verena C.; Stetter, Karl O. A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont. Nature. 2002-05-02, 417 (6884) [2022-10-13]. Bibcode:2002Natur.417...63H. ISSN 0028-0836. PMID 11986665. doi:10.1038/417063a. (原始内容存档于2022-10-18) (英语).
- ^ Barns, S M; Delwiche, C F; Palmer, J D; Pace, N R. Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences.. Proceedings of the National Academy of Sciences. 1996-08-20, 93 (17). Bibcode:1996PNAS...93.9188B. ISSN 0027-8424. PMC 38617 . PMID 8799176. doi:10.1073/pnas.93.17.9188 (英语).
- ^ Elkins, James G.; Podar, Mircea; Graham, David E.; Makarova, Kira S.; Wolf, Yuri; Randau, Lennart; Hedlund, Brian P.; Brochier-Armanet, Céline; Kunin, Victor; Anderson, Iain; Lapidus, Alla. A korarchaeal genome reveals insights into the evolution of the Archaea. Proceedings of the National Academy of Sciences. 2008-06-10, 105 (23) [2022-10-13]. Bibcode:2008PNAS..105.8102E. ISSN 0027-8424. PMC 2430366 . PMID 18535141. doi:10.1073/pnas.0801980105. (原始内容存档于2022-11-08) (英语).
- ^ Baker, Brett J.; Tyson, Gene W.; Webb, Richard I.; Flanagan, Judith; Hugenholtz, Philip; Allen, Eric E.; Banfield, Jillian F. Lineages of Acidophilic Archaea Revealed by Community Genomic Analysis. Science. 2006-12-22, 314 (5807) [2022-10-13]. Bibcode:2006Sci...314.1933B. ISSN 0036-8075. PMID 17185602. doi:10.1126/science.1132690. (原始内容存档于2022-10-14) (英语).
- ^ 22.0 22.1 Baker BJ; Comolli LR; Dick GJ; et al. Enigmatic, ultrasmall, uncultivated Archaea. Proceedings of the National Academy of Sciences of the United States of America. May 2010, 107 (19): 8806–11. Bibcode:2010PNAS..107.8806B. PMC 2889320 . PMID 20421484. doi:10.1073/pnas.0914470107.
- ^ Guy, Lionel; Ettema, Thijs J. G. The archaeal ‘TACK’ superphylum and the origin of eukaryotes. Trends in Microbiology. 2011-12-01, 19 (12) [2022-10-13]. ISSN 0966-842X. PMID 22018741. doi:10.1016/j.tim.2011.09.002. (原始内容存档于2013-10-16) (英语).
- ^ Zaremba-Niedzwiedzka, Katarzyna; Caceres, Eva F.; Saw, Jimmy H.; Bäckström, Disa; Juzokaite, Lina; Vancaester, Emmelien; Seitz, Kiley W.; Anantharaman, Karthik; Starnawski, Piotr; Kjeldsen, Kasper U.; Stott, Matthew B. Asgard archaea illuminate the origin of eukaryotic cellular complexity. Nature. 2017-01-19, 541 (7637) [2022-10-13]. ISSN 0028-0836. doi:10.1038/nature21031. (原始内容存档于2022-12-03) (英语).
- ^ Talbert, Paul B.; Henikoff, Steven. Histone variants — ancient wrap artists of the epigenome. Nature Reviews Molecular Cell Biology. 2010-04, 11 (4) [2022-10-13]. ISSN 1471-0080. doi:10.1038/nrm2861. (原始内容存档于2022-10-17) (英语).
- ^ Sandman, Kathleen; Reeve, John N. Archaeal histones and the origin of the histone fold. Current Opinion in Microbiology. Antimicrobials/Genomics. 2006-10-01, 9 (5). ISSN 1369-5274. doi:10.1016/j.mib.2006.08.003 (英语).
- ^ 27.0 27.1 27.2 Zillig, Wolfram. Comparative biochemistry of Archaea and Bacteria. Current Opinion in Genetics & Development. 1991-12-01, 1 (4) [2022-10-13]. ISSN 0959-437X. doi:10.1016/S0959-437X(05)80206-0. (原始内容存档于2020-07-01) (英语).
- ^ Bell, Stephen D; Jackson, Stephen P. Mechanism and regulation of transcription in archaea. Current Opinion in Microbiology. 2001-04-01, 4 (2). ISSN 1369-5274. PMID 11282478. doi:10.1016/S1369-5274(00)00190-9 (英语).
- ^ Reeve, John N. Archaeal chromatin and transcription. Molecular Microbiology. 2003-05, 48 (3) [2022-10-13]. ISSN 0950-382X. PMID 12694606. doi:10.1046/j.1365-2958.2003.03439.x. (原始内容存档于2022-10-01).
- ^ Kelman, Lori M.; Kelman, Zvi. Archaea: an archetype for replication initiation studies?. Molecular Microbiology. 2003-05, 48 (3) [2022-10-13]. ISSN 0950-382X. PMID 12694608. doi:10.1046/j.1365-2958.2003.03369.x. (原始内容存档于2022-10-18).
- ^ Phillips, Gabriela; Chikwana, Vimbai M.; Maxwell, Adrienne; El-Yacoubi, Basma; Swairjo, Manal A.; Iwata-Reuyl, Dirk; Crécy-Lagard, Valérie de. Discovery and Characterization of an Amidinotransferase Involved in the Modification of Archaeal tRNA *♦. Journal of Biological Chemistry. 2010-04-23, 285 (17). ISSN 0021-9258. PMC 2857094 . PMID 20129918. doi:10.1074/jbc.M110.102236 (英语).
- ^ Hara, Futoshi; Yamashiro, Kan; Nemoto, Naoki; Ohta, Yoshinori; Yokobori, Shin-ichi; Yasunaga, Takuo; Hisanaga, Shin-ichi; Yamagishi, Akihiko. An Actin Homolog of the Archaeon Thermoplasma acidophilum That Retains the Ancient Characteristics of Eukaryotic Actin. Journal of Bacteriology. 2007-03, 189 (5) [2022-10-13]. ISSN 0021-9193. PMC 1855749 . PMID 17189356. doi:10.1128/JB.01454-06. (原始内容存档于2023-01-23) (英语).
- ^ Trent, Jonathan D.; Kagawa, Hiromi K.; Yaoi, Takuro; Olle, Eric; Zaluzec, Nestor J. Chaperonin filaments: The archaeal cytoskeleton?. Proceedings of the National Academy of Sciences. 1997-05-13, 94 (10) [2022-10-13]. Bibcode:1997PNAS...94.5383T. ISSN 0027-8424. PMC 24687 . PMID 9144246. doi:10.1073/pnas.94.10.5383. (原始内容存档于2022-10-16) (英语).
- ^ Hixon, William G.; Searcy, Dennis G. Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts. Biosystems. 1993-01-01, 29 (2). ISSN 0303-2647. PMID 8374067. doi:10.1016/0303-2647(93)90091-P (英语).
- ^ Woese, C R. There must be a prokaryote somewhere: microbiology's search for itself. Microbiological Reviews. 1994-03, 58 (1) [2022-10-13]. ISSN 0146-0749. PMC 372949 . PMID 8177167. doi:10.1128/mr.58.1.1-9.1994. (原始内容存档于2022-10-18) (英语).
- ^ 36.0 36.1 Thomas, Nikhil A.; Bardy, Sonia L.; Jarrell, Ken F. The archaeal flagellum: a different kind of prokaryotic motility structure. FEMS Microbiology Reviews. 2001-04, 25 (2) [2022-10-13]. ISSN 1574-6976. PMID 11250034. doi:10.1111/j.1574-6976.2001.tb00575.x. (原始内容存档于2022-11-21) (英语).
- ^ Golyshina, O V; Pivovarova, T A; Karavaiko, G I; Kondratéva, T F; Moore, E R; Abraham, W R; Lünsdorf, H; Timmis, K N; Yakimov, M M; Golyshin, P N. Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea.. International Journal of Systematic and Evolutionary Microbiology. 2000-05-01, 50 (3) [2022-10-13]. ISSN 1466-5026. PMID 10843038. doi:10.1099/00207713-50-3-997. (原始内容存档于2023-01-23) (英语).
- ^ Engelhardt, Harald; Peters, Jürgen. Structural Research on Surface Layers: A Focus on Stability, Surface Layer Homology Domains, and Surface Layer–Cell Wall Interactions. Journal of Structural Biology. 1998-12-15, 124 (2) [2022-10-13]. ISSN 1047-8477. PMID 10049812. doi:10.1006/jsbi.1998.4070. (原始内容存档于2020-10-02) (英语).
- ^ Kandler, O.; König, H. Cell wall polymers in Archaea (Archaebacteria). Cellular and Molecular Life Sciences CMLS. 1998-04, 54 (4). ISSN 1420-682X. doi:10.1007/s000180050156 (英语).
- ^ Howland, John L. The Surprising Archaea: Discovering Another Domain of Life. Oxford: Oxford University Press. 2000: 32. ISBN 0-19-511183-4.
- ^ Gophna, Uri; Ron, Eliora Z.; Graur, Dan. Bacterial type III secretion systems are ancient and evolved by multiple horizontal-transfer events. Gene. 2003-07-17, 312 [2022-10-13]. ISSN 0378-1119. PMID 12909351. doi:10.1016/S0378-1119(03)00612-7. (原始内容存档于2018-11-06) (英语).
- ^ Nguyen L, Paulsen IT, Tchieu J, Hueck CJ, Saier MH. Phylogenetic analyses of the constituents of Type III protein secretion systems. J. Mol. Microbiol. Biotechnol. April 2000, 2 (2): 125–44. PMID 10939240.
- ^ Ng, Sandy Y.M.; Chaban, Bonnie; Jarrell, Ken F. Archaeal Flagella, Bacterial Flagella and Type IV Pili: A Comparison of Genes and Posttranslational Modifications. Microbial Physiology. 2006, 11 (3-5) [2022-10-13]. ISSN 2673-1665. PMID 16983194. doi:10.1159/000094053. (原始内容存档于2014-04-21) (英语).
- ^ Bardy, Sonia L.; Ng, Sandy Y. M.; Jarrell, Ken F. Prokaryotic motility structures. Microbiology. 2003-02-01, 149 (2) [2022-10-13]. ISSN 1350-0872. PMID 12624192. doi:10.1099/mic.0.25948-0. (原始内容存档于2022-10-15) (英语).
- ^ 45.0 45.1 Allers, Thorsten; Mevarech, Moshe. Archaeal genetics — the third way. Nature Reviews Genetics. 2005-01, 6 (1) [2022-10-14]. ISSN 1471-0056. PMID 15630422. doi:10.1038/nrg1504. (原始内容存档于2022-10-16) (英语).
- ^ Galagan, James E.; Nusbaum, Chad; Roy, Alice; Endrizzi, Matthew G.; Macdonald, Pendexter; FitzHugh, Will; Calvo, Sarah; Engels, Reinhard; Smirnov, Serge; Atnoor, Deven; Brown, Adam. The Genome of M. acetivorans Reveals Extensive Metabolic and Physiological Diversity. Genome Research. 2002-04-01, 12 (4). ISSN 1088-9051. PMC 187521 . PMID 11932238. doi:10.1101/gr.223902 (英语).
- ^ Waters, Elizabeth; Hohn, Michael J.; Ahel, Ivan; Graham, David E.; Adams, Mark D.; Barnstead, Mary; Beeson, Karen Y.; Bibbs, Lisa; Bolanos, Randall; Keller, Martin; Kretz, Keith. The genome of Nanoarchaeum equitans: Insights into early archaeal evolution and derived parasitism. Proceedings of the National Academy of Sciences. 2003-10-28, 100 (22) [2022-10-14]. Bibcode:2003PNAS..10012984W. ISSN 0027-8424. PMC 240731 . PMID 14566062. doi:10.1073/pnas.1735403100. (原始内容存档于2022-10-18) (英语).
- ^ Schleper, C; Holz, I; Janekovic, D; Murphy, J; Zillig, W. A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating. Journal of Bacteriology. 1995-08, 177 (15) [2022-10-14]. ISSN 0021-9193. PMC 177192 . PMID 7635827. doi:10.1128/jb.177.15.4417-4426.1995. (原始内容存档于2022-10-18) (英语).
- ^ Sota M; Top EM. Horizontal Gene Transfer Mediated by Plasmids. Plasmids: Current Research and Future Trends. Caister Academic Press. 2008 [2014-01-15]. ISBN 978-1-904455-35-6. (原始内容存档于2008-04-11).
- ^ Xiang, Xiaoyu; Chen, Lanming; Huang, Xiaoxing; Luo, Yuanmin; She, Qunxin; Huang, Li. Sulfolobus tengchongensis Spindle-Shaped Virus STSV1: Virus-Host Interactions and Genomic Features. Journal of Virology. 2005-07, 79 (14). ISSN 0022-538X. PMC 1168784 . PMID 15994761. doi:10.1128/JVI.79.14.8677-8686.2005 (英语).
- ^ Prangishvili, David; Forterre, Patrick; Garrett, Roger A. Viruses of the Archaea: a unifying view. Nature Reviews Microbiology. 2006-11-01, 4 (11) [2022-10-14]. ISSN 1740-1526. PMID 17041631. doi:10.1038/nrmicro1527. (原始内容存档于2022-10-17) (英语).
- ^ Prangishvili, D.; Garrett, R.A. Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses. Biochemical Society Transactions. 2004-04-01, 32 (2) [2022-10-14]. ISSN 0300-5127. PMID 15046572. doi:10.1042/bst0320204. (原始内容存档于2023-01-19) (英语).
- ^ Pietilä MK, Roine E, Paulin L, Kalkkinen N, Bamford DH. An ssDNA virus infecting archaea; A new lineage of viruses with a membrane envelope. Mol. Microbiol. March 2009, 72 (2): 307–19. PMID 19298373. doi:10.1111/j.1365-2958.2009.06642.x.
- ^ Mochizuki, Tomohiro; Krupovic, Mart; Pehau-Arnaudet, Gérard; Sako, Yoshihiko; Forterre, Patrick; Prangishvili, David. Archaeal virus with exceptional virion architecture and the largest single-stranded DNA genome. Proceedings of the National Academy of Sciences. 2012-08-14, 109 (33) [2022-10-14]. Bibcode:2012PNAS..10913386M. ISSN 0027-8424. PMC 3421227 . PMID 22826255. doi:10.1073/pnas.1203668109. (原始内容存档于2022-10-21) (英语).
- ^ Mojica, Francisco J.M.; Díez-Villaseñor, Chc)sar; García-Martínez, Jesús; Soria, Elena. Intervening Sequences of Regularly Spaced Prokaryotic Repeats Derive from Foreign Genetic Elements. Journal of Molecular Evolution. 2005-02, 60 (2). ISSN 0022-2844. PMID 15791728. doi:10.1007/s00239-004-0046-3 (英语).
- ^ Makarova, Kira S; Grishin, Nick V; Shabalina, Svetlana A; Wolf, Yuri I; Koonin, Eugene V. A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biology Direct. 2006-12, 1 (1) [2022-10-14]. ISSN 1745-6150. PMC 1462988 . PMID 16545108. doi:10.1186/1745-6150-1-7. (原始内容存档于2022-11-09) (英语).
- ^ Graham, David E.; Overbeek, Ross; Olsen, Gary J.; Woese, Carl R. An archaeal genomic signature. Proceedings of the National Academy of Sciences. 2000-03-28, 97 (7) [2022-10-14]. Bibcode:2000PNAS...97.3304G. ISSN 0027-8424. PMC 16234 . PMID 10716711. doi:10.1073/pnas.97.7.3304. (原始内容存档于2022-10-14) (英语).
- ^ Gaasterland, Terry. Archaeal genomics. Current Opinion in Microbiology. 1999-10-01, 2 (5) [2022-10-14]. ISSN 1369-5274. PMID 10508726. doi:10.1016/S1369-5274(99)00014-4. (原始内容存档于2019-03-22) (英语).
- ^ Werner, Finn. Structure and function of archaeal RNA polymerases. Molecular Microbiology. 2007-09, 65 (6) [2022-10-14]. ISSN 0950-382X. PMID 17697097. doi:10.1111/j.1365-2958.2007.05876.x. (原始内容存档于2022-10-17) (英语).
- ^ Aravind, L. DNA-binding proteins and evolution of transcription regulation in the archaea. Nucleic Acids Research. 1999-12-01, 27 (23) [2022-10-14]. PMC 148756 . PMID 10556324. doi:10.1093/nar/27.23.4658. (原始内容存档于2022-08-08).
- ^ Lykke-Andersen J, Aagaard C, Semionenkov M, Garrett RA. Archaeal introns: splicing, intercellular mobility and evolution. Trends Biochem. Sci. September 1997, 22 (9): 326–31. PMID 9301331. doi:10.1016/S0968-0004(97)01113-4.
- ^ Watanabe, Yoh-ichi; Yokobori, Shin-ichi; Inaba, Toshiro; Yamagishi, Akihiko; Oshima, Tairo; Kawarabayasi, Yutaka; Kikuchi, Hisasi; Kita, Kiyoshi. Introns in protein-coding genes in Archaea. FEBS Letters. 2002-01-02, 510 (1-2). PMID 11755525. doi:10.1016/S0014-5793(01)03219-7 (英语).
- ^ Yoshinari, Shigeo; Itoh, Takashi; Hallam, Steven J.; DeLong, Edward F.; Yokobori, Shin-ichi; Yamagishi, Akihiko; Oshima, Tairo; Kita, Kiyoshi; Watanabe, Yoh-ichi. Archaeal pre-mRNA splicing: A connection to hetero-oligomeric splicing endonuclease. Biochemical and Biophysical Research Communications. 2006-08-04, 346 (3) [2022-10-14]. ISSN 0006-291X. PMID 16781672. doi:10.1016/j.bbrc.2006.06.011. (原始内容存档于2019-03-22) (英语).
- ^ Genome List - Genome. NCBI. [2005-07-13]. (原始内容存档于2021-02-10).
- ^ Rosenshine, Ilan; Tchelet, Ronen; Mevarech, Moshe. The Mechanism of DNA Transfer in the Mating System of an Archaebacterium. Science. 1989-09-22, 245 (4924) [2022-10-14]. ISSN 0036-8075. PMID 2818746. doi:10.1126/science.2818746. (原始内容存档于2022-10-14) (英语).
- ^ 66.0 66.1 66.2 Fröls, Sabrina; Ajon, Malgorzata; Wagner, Michaela; Teichmann, Daniela; Zolghadr, Behnam; Folea, Mihaela; Boekema, Egbert J.; Driessen, Arnold J. M.; Schleper, Christa; Albers, Sonja-Verena. UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation: UV-inducible cellular aggregation. Molecular Microbiology. 2008-10-09, 70 (4) [2022-10-14]. PMID 2818746. doi:10.1111/j.1365-2958.2008.06459.x. (原始内容存档于2022-10-14) (英语).
- ^ 67.0 67.1 67.2 Ajon, Małgorzata; Fröls, Sabrina; van Wolferen, Marleen; Stoecker, Kilian; Teichmann, Daniela; Driessen, Arnold J. M.; Grogan, Dennis W.; Albers, Sonja-Verena; Schleper, Christa. UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili: UV-inducible DNA exchange in hyperthermophilic archaea. Molecular Microbiology. 2011-11, 82 (4) [2022-10-14]. PMID 21999488. doi:10.1111/j.1365-2958.2011.07861.x. (原始内容存档于2022-10-14) (英语).
- ^ Fröls, Sabrina; White, Malcolm F.; Schleper, Christa. Reactions to UV damage in the model archaeon Sulfolobus solfataricus. Biochemical Society Transactions. 2009-02-01, 37 (1) [2022-10-14]. ISSN 0300-5127. PMID 19143598. doi:10.1042/BST0370036. (原始内容存档于2022-10-17) (英语).
- ^ Bernstein, Harris; Bernstein, Carol. Evolutionary Origin and Adaptive Function of Meiosis. IntechOpen. 2013-09-11. ISBN 978-953-51-1197-9 (英语).[失效链接]
- ^ Krieg, Noel. Bergey's Manual of Systematic Bacteriology. US: Springer. 2005: 21–6. ISBN 978-0-387-24143-2.
- ^ 71.0 71.1 Bernander, Rolf. Archaea and the cell cycle. Molecular Microbiology. 1998-08, 29 (4). PMID 9767564. doi:10.1046/j.1365-2958.1998.00956.x (英语).
- ^ Kelman, Lori M.; Kelman, Zvi. Multiple origins of replication in archaea. Trends in Microbiology. 2004-09-01, 12 (9) [2022-10-14]. ISSN 0966-842X. PMID 15337158. doi:10.1016/j.tim.2004.07.001. (原始内容存档于2013-10-16) (英语).
- ^ Onyenwoke, RobU.; Brill, JuliaA.; Farahi, Kamyar; Wiegel, Juergen. Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch (Firmicutes). Archives of Microbiology. 2004-10, 182 (2-3). ISSN 0302-8933. PMID 15340788. doi:10.1007/s00203-004-0696-y (英语).
- ^ Kostrikina, N. A.; Zvyagintseva, I. S.; Duda, V. I. Cytological peculiarities of some extremely halophilic soil archaeobacteria. Archives of Microbiology. 1991-10, 156 (5). ISSN 0302-8933. doi:10.1007/BF00248708 (英语).
- ^ DeLong, Edward F.; Pace, Norman R. Environmental Diversity of Bacteria and Archaea. Systematic Biology. 2001-08-01, 50 (4). PMID 12116647. doi:10.1080/106351501750435040.
- ^ 76.0 76.1 Valentine DL. Adaptations to energy stress dictate the ecology and evolution of the Archaea. Nature Reviews Microbiology. 2007, 5 (4): 316–23. PMID 17334387. doi:10.1038/nrmicro1619.
- ^ 77.0 77.1 Pikuta, Elena V.; Hoover, Richard B.; Tang, Jane. Microbial Extremophiles at the Limits of Life. Critical Reviews in Microbiology. 2007-01, 33 (3) [2022-10-14]. ISSN 1040-841X. PMID 17653987. doi:10.1080/10408410701451948. (原始内容存档于2022-06-19) (英语).
- ^ Madigan MT, Martino JM. Brock Biology of Microorganisms 11th. Pearson. 2006: 136. ISBN 0-13-196893-9.
- ^ Takai, Ken; Nakamura, Kentaro; Toki, Tomohiro; Tsunogai, Urumu; Miyazaki, Masayuki; Miyazaki, Junichi; Hirayama, Hisako; Nakagawa, Satoshi; Nunoura, Takuro; Horikoshi, Koki. Cell proliferation at 122°C and isotopically heavy CH 4 production by a hyperthermophilic methanogen under high-pressure cultivation. Proceedings of the National Academy of Sciences. 2008-08-05, 105 (31) [2022-10-14]. Bibcode:2008PNAS..10510949T. ISSN 0027-8424. PMC 2490668 . PMID 18664583. doi:10.1073/pnas.0712334105. (原始内容存档于2022-10-21) (英语).
- ^ Ciaramella, Maria; Napoli, Alessandra; Rossi, Mosè. Another extreme genome: how to live at pH 0. Trends in Microbiology. 2005-02-01, 13 (2). ISSN 0966-842X. PMID 15680761. doi:10.1016/j.tim.2004.12.001 (英语).
- ^ Javaux, Emmanuelle J. Extreme life on Earth—past, present and possibly beyond. Research in Microbiology. Space Microbiology. 2006-01-01, 157 (1) [2022-10-14]. ISSN 0923-2508. PMID 16376523. doi:10.1016/j.resmic.2005.07.008. (原始内容存档于2020-10-31) (英语).
- ^ Nealson, K. H. Post-Viking microbiology: new approaches, new data, new insights. Origins of Life and Evolution of the Biosphere: The Journal of the International Society for the Study of the Origin of Life. 1999-01, 29 (1) [2022-10-14]. ISSN 0169-6149. PMID 11536899. doi:10.1023/a:1006515817767. (原始内容存档于2022-10-05).
- ^ Davies, P. C. W. The Transfer of Viable Microorganisms Between Planets. Bock, Gregoy R. (编). Novartis Foundation Symposia. Chichester, UK: John Wiley & Sons, Ltd. 2007-09-28: 304–317 [2022-10-14]. ISBN 978-0-470-51498-6. PMID 9243022. doi:10.1002/9780470514986.ch16. (原始内容存档于2022-10-16).
- ^ López-García, P. Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front. FEMS Microbiology Ecology. 2001-07, 36 (2-3). PMID 11451524. doi:10.1016/S0168-6496(01)00133-7.
- ^ Karner, Markus B.; DeLong, Edward F.; Karl, David M. Archaeal dominance in the mesopelagic zone of the Pacific Ocean. Nature. 2001-01, 409 (6819) [2022-10-14]. ISSN 0028-0836. PMID 11206545. doi:10.1038/35054051. (原始内容存档于2022-12-09) (英语).
- ^ Giovannoni, Stephen J.; Stingl, Ulrich. Molecular diversity and ecology of microbial plankton. Nature. 2005-09, 437 (7057) [2022-10-14]. Bibcode:2005Natur.437..343G. ISSN 0028-0836. PMID 16163344. doi:10.1038/nature04158. (原始内容存档于2023-01-23) (英语).
- ^ DeLong, Edward F.; Karl, David M. Genomic perspectives in microbial oceanography. Nature. 2005-09-15, 437 (7057) [2022-10-14]. Bibcode:2005Natur.437..336D. ISSN 0028-0836. PMID 16163343. doi:10.1038/nature04157. (原始内容存档于2023-01-23) (英语).
- ^ Könneke, Martin; Bernhard, Anne E.; de la Torre, José R.; Walker, Christopher B.; Waterbury, John B.; Stahl, David A. Isolation of an autotrophic ammonia-oxidizing marine archaeon. Nature. 2005-09, 437 (7058) [2022-10-14]. Bibcode:2005Natur.437..543K. ISSN 0028-0836. PMID 16177789. doi:10.1038/nature03911. (原始内容存档于2022-10-20) (英语).
- ^ Agogué, Hélène; Brink, Maaike; Dinasquet, Julie; Herndl, Gerhard J. Major gradients in putatively nitrifying and non-nitrifying Archaea in the deep North Atlantic. Nature. 2008-12-11, 456 (7223) [2022-10-14]. Bibcode:2008Natur.456..788A. ISSN 0028-0836. PMID 19037244. doi:10.1038/nature07535. (原始内容存档于2022-10-18) (英语).
- ^ Lipp, Julius S.; Morono, Yuki; Inagaki, Fumio; Hinrichs, Kai-Uwe. Significant contribution of Archaea to extant biomass in marine subsurface sediments. Nature. 2008-08, 454 (7207) [2022-10-14]. Bibcode:2008Natur.454..991L. ISSN 0028-0836. PMID 18641632. doi:10.1038/nature07174. (原始内容存档于2022-11-02) (英语).
- ^ Cabello, Purificación; Roldán, M. Dolores; Moreno-Vivián, Conrado. Nitrate reduction and the nitrogen cycle in archaea. Microbiology. 2004-11-01, 150 (11) [2022-10-14]. ISSN 1350-0872. PMID 15528644. doi:10.1099/mic.0.27303-0. (原始内容存档于2022-10-15) (英语).
- ^ Mehta MP, Baross JA. Nitrogen fixation at 92 degrees C by a hydrothermal vent archaeon. Science. December 2006, 314 (5806): 1783–6. Bibcode:2006Sci...314.1783M. PMID 17170307. doi:10.1126/science.1134772.
- ^ Francis CA, Beman JM, Kuypers MM. New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation. ISME J. May 2007, 1 (1): 19–27. PMID 18043610. doi:10.1038/ismej.2007.8.
- ^ Coolen, Marco J. L.; Abbas, Ben; van Bleijswijk, Judith; Hopmans, Ellen C.; Kuypers, Marcel M. M.; Wakeham, Stuart G.; Sinninghe Damsté, Jaap S. Putative ammonia-oxidizing Crenarchaeota in suboxic waters of the Black Sea: a basin-wide ecological study using 16S ribosomal and functional genes and membrane lipids. Environmental Microbiology. 2007-04, 9 (4). ISSN 1462-2912. PMID 17359272. doi:10.1111/j.1462-2920.2006.01227.x (英语).
- ^ Leininger, S.; Urich, T.; Schloter, M.; Schwark, L.; Qi, J.; Nicol, G. W.; Prosser, J. I.; Schuster, S. C.; Schleper, C. Archaea predominate among ammonia-oxidizing prokaryotes in soils. Nature. 2006-08, 442 (7104) [2022-10-14]. Bibcode:2006Natur.442..806L. ISSN 0028-0836. PMID 16915287. doi:10.1038/nature04983. (原始内容存档于2022-10-18) (英语).
- ^ Baker, Brett J.; Banfield, Jillian F. Microbial communities in acid mine drainage. FEMS microbiology ecology. 2003-05-01, 44 (2) [2022-10-14]. ISSN 1574-6941. PMID 19719632. doi:10.1016/S0168-6496(03)00028-X. (原始内容存档于2022-10-16).
- ^ Schimel, Joshua. Playing scales in the methane cycle: From microbial ecology to the globe. Proceedings of the National Academy of Sciences. 2004-08-24, 101 (34) [2022-10-14]. Bibcode:2004PNAS..10112400S. ISSN 0027-8424. PMC 515073 . PMID 15314221. doi:10.1073/pnas.0405075101. (原始内容存档于2022-10-18) (英语).
- ^ Trace Gases: Current Observations, Trends, and Budgets. Climate Change 2001. United Nations Environment Programme. [2014-09-08]. (原始内容存档于2011-12-10).
- ^ IPCC AR5 WG1. Climate Change 2013: The Physical Science Basis - Summary for Policymakers (PDF). Cambridge University Press. 2013 [2014-09-08]. (原始内容存档 (PDF)于2014-02-26).
- ^ IPCC AR5 WG1. Climate Change 2013: The Physical Science Basis - Anthropogenic and Natural Radiative Forcing Supplementary Material (PDF). Cambridge University Press. 2013 [2014-09-08]. (原始内容存档 (PDF)于2016-03-04).
- ^ Eckburg, Paul B.; Lepp, Paul W.; Relman, David A. Archaea and Their Potential Role in Human Disease. Infection and Immunity. 2003-02, 71 (2) [2022-10-14]. ISSN 0019-9567. PMC 145348 . PMID 12540534. doi:10.1128/IAI.71.2.591-596.2003. (原始内容存档于2023-01-23) (英语).
- ^ Cavicchioli R, Curmi P, Saunders N, Thomas T. Pathogenic archaea: do they exist?. BioEssays. 2003, 25 (11): 1119–28. PMID 14579252. doi:10.1002/bies.10354.
- ^ Lepp, Paul W.; Brinig, Mary M.; Ouverney, Cleber C.; Palm, Katherine; Armitage, Gary C.; Relman, David A. Methanogenic Archaea and human periodontal disease. Proceedings of the National Academy of Sciences. 2004-04-20, 101 (16) [2022-10-14]. Bibcode:2004PNAS..101.6176L. ISSN 0027-8424. PMC 395942 . PMID 15067114. doi:10.1073/pnas.0308766101. (原始内容存档于2022-10-14) (英语).
- ^ Vianna, M. E.; Conrads, G.; Gomes, B. P. F. A.; Horz, H. P. Identification and Quantification of Archaea Involved in Primary Endodontic Infections. Journal of Clinical Microbiology. 2006-04, 44 (4) [2022-10-14]. ISSN 0095-1137. PMC 1448633 . PMID 16597851. doi:10.1128/JCM.44.4.1274-1282.2006. (原始内容存档于2023-01-23) (英语).
- ^ Waters, Elizabeth; Hohn, Michael J.; Ahel, Ivan; Graham, David E.; Adams, Mark D.; Barnstead, Mary; Beeson, Karen Y.; Bibbs, Lisa; Bolanos, Randall; Keller, Martin; Kretz, Keith. The genome of Nanoarchaeum equitans: Insights into early archaeal evolution and derived parasitism. Proceedings of the National Academy of Sciences. 2003-10-28, 100 (22) [2022-10-14]. ISSN 0027-8424. PMC 240731 . PMID 14566062. doi:10.1073/pnas.1735403100. (原始内容存档于2022-10-18) (英语).
- ^ Jahn, Ulrike; Gallenberger, Martin; Paper, Walter; Junglas, Benjamin; Eisenreich, Wolfgang; Stetter, Karl O.; Rachel, Reinhard; Huber, Harald. Nanoarchaeum equitans and Ignicoccus hospitalis : New Insights into a Unique, Intimate Association of Two Archaea. Journal of Bacteriology. 2008-03, 190 (5) [2022-10-14]. ISSN 0021-9193. PMC 2258681 . PMID 18165302. doi:10.1128/JB.01731-07. (原始内容存档于2023-01-23) (英语).
- ^ Baker, Brett J.; Comolli, Luis R.; Dick, Gregory J.; Hauser, Loren J.; Hyatt, Doug; Dill, Brian D.; Land, Miriam L.; VerBerkmoes, Nathan C.; Hettich, Robert L.; Banfield, Jillian F. Enigmatic, ultrasmall, uncultivated Archaea. Proceedings of the National Academy of Sciences. 2010-05-11, 107 (19) [2022-10-14]. Bibcode:2010PNAS..107.8806B. ISSN 0027-8424. PMC 2889320 . PMID 20421484. doi:10.1073/pnas.0914470107. (原始内容存档于2022-10-21) (英语).
- ^ Chaban, Bonnie; Ng, Sandy Y.M; Jarrell, Ken F. Archaeal habitats — from the extreme to the ordinary. Canadian Journal of Microbiology. 2006-02-01, 52 (2) [2022-10-14]. ISSN 0008-4166. PMID 16541146. doi:10.1139/w05-147. (原始内容存档于2019-05-18) (英语).
- ^ Schink, B. Energetics of syntrophic cooperation in methanogenic degradation. Microbiology and Molecular Biology Reviews. 1997-06, 61 (2) [2022-10-14]. ISSN 1092-2172. PMC 232610 . PMID 9184013. doi:10.1128/mmbr.61.2.262-280.1997. (原始内容存档于2022-10-14) (英语).
- ^ Lange, Marianne; Westermann, Peter; Ahring, Birgitte Kiær. Archaea in protozoa and metazoa. Applied Microbiology and Biotechnology. 2005-02, 66 (5). ISSN 0175-7598. PMID 15630514. doi:10.1007/s00253-004-1790-4 (英语).
- ^ van Hoek, Angela H. A. M.; van Alen, Theo A.; Sprakel, Vera S. I.; Leunissen, Jack A. M.; Brigge, Theo; Vogels, Godfried D.; Hackstein, Johannes H. P. Multiple Acquisition of Methanogenic Archaeal Symbionts by Anaerobic Ciliates. Molecular Biology and Evolution. 2000-02-01, 17 (2) [2022-10-14]. ISSN 0737-4038. PMID 10677847. doi:10.1093/oxfordjournals.molbev.a026304. (原始内容存档于2023-01-23) (英语).
- ^ Preston, C M; Wu, K Y; Molinski, T F; DeLong, E F. A psychrophilic crenarchaeon inhabits a marine sponge: Cenarchaeum symbiosum gen. nov., sp. nov.. Proceedings of the National Academy of Sciences. 1996-06-25, 93 (13). Bibcode:1996PNAS...93.6241P. ISSN 0027-8424. PMC 39006 . PMID 8692799. doi:10.1073/pnas.93.13.6241 (英语).
- ^ Eckburg, Paul B.; Bik, Elisabeth M.; Bernstein, Charles N.; Purdom, Elizabeth; Dethlefsen, Les; Sargent, Michael; Gill, Steven R.; Nelson, Karen E.; Relman, David A. Diversity of the Human Intestinal Microbial Flora. Science. 2005-06-10, 308 (5728) [2022-10-14]. Bibcode:2005Sci...308.1635E. ISSN 0036-8075. PMC 1395357 . PMID 15831718. doi:10.1126/science.1110591. (原始内容存档于2023-03-11) (英语).
- ^ Samuel, Buck S.; Gordon, Jeffrey I. A humanized gnotobiotic mouse model of host–archaeal–bacterial mutualism. Proceedings of the National Academy of Sciences. 2006-06-27, 103 (26) [2022-10-14]. Bibcode:2006PNAS..10310011S. ISSN 0027-8424. PMC 1479766 . PMID 16782812. doi:10.1073/pnas.0602187103. (原始内容存档于2022-10-20) (英语).
- ^ Wegley, L; Yu, Y; Breitbart, M; Casas, V; Kline, Di; Rohwer, F. Coral-associated Archaea. Marine Ecology Progress Series. 2004, 273 [2022-10-14]. ISSN 0171-8630. doi:10.3354/meps273089. (原始内容存档于2022-10-18) (英语).
- ^ Chelius, M.K.; Triplett, E.W. The Diversity of Archaea and Bacteria in Association with the Roots of Zea mays L.. Microbial Ecology. 2001-04, 41 (3). ISSN 0095-3628. PMID 11391463. doi:10.1007/s002480000087 (英语).
- ^ Simon, Holly M.; Dodsworth, Jeremy A.; Goodman, Robert M. Crenarchaeota colonize terrestrial plant roots. Environmental Microbiology. 2000-10, 2 (5). PMID 11233158. doi:10.1046/j.1462-2920.2000.00131.x (英语).
- ^ Breithaupt, Holger. The hunt for living gold: The search for organisms in extreme environments yields useful enzymes for industry. EMBO reports. 2001-11, 2 (11). ISSN 1469-221X. PMC 1084137 . PMID 11713183. doi:10.1093/embo-reports/kve238 (英语).
- ^ 119.0 119.1 Egorova, Ksenia; Antranikian, Garabed. Industrial relevance of thermophilic Archaea. Current Opinion in Microbiology. Growth development / edited by John N Reeve and Ruth A Schmitz. 2005-12-01, 8 (6) [2022-10-14]. ISSN 1369-5274. PMID 16257257. doi:10.1016/j.mib.2005.10.015. (原始内容存档于2011-12-16) (英语).
- ^ Synowiecki, Józef; Grzybowska, Beata; Zdziebło, Anna. Sources, Properties and Suitability of New Thermostable Enzymes in Food Processing. Critical Reviews in Food Science and Nutrition. 2006-04, 46 (3) [2022-10-14]. ISSN 1040-8398. PMID 16527752. doi:10.1080/10408690590957296. (原始内容存档于2022-08-08) (英语).
- ^ Jenney Jr, Francis E.; Adams, Michael W. W. The impact of extremophiles on structural genomics (and vice versa). Extremophiles. 2008-01, 12 (1). ISSN 1431-0651. PMID 17563834. doi:10.1007/s00792-007-0087-9 (英语).
- ^ Schiraldi, Chiara; Giuliano, Mariateresa; De Rosa, Mario. Perspectives on biotechnological applications of archaea. Archaea. 2002, 1 (2) [2022-10-14]. ISSN 1472-3646. PMC 2685559 . PMID 15803645. doi:10.1155/2002/436561. (原始内容存档于2019-05-18) (英语).
- ^ Norris, P. R.; Burton, N. P.; Foulis, N. A. Acidophiles in bioreactor mineral processing. Extremophiles: Life Under Extreme Conditions. 2000-04, 4 (2) [2022-10-14]. ISSN 1431-0651. PMID 10805560. doi:10.1007/s007920050139. (原始内容存档于2022-10-14).
- ^ Shand RF; Leyva KJ. Archaeal Antimicrobials: An Undiscovered Country. Blum P (ed.) (编). Archaea: New Models for Prokaryotic Biology. Caister Academic Press. 2008. ISBN 978-1-904455-27-1.
- ^ Domain Archaea. LPSN. [2024-05-17].
- ^ Kato S, Ogasawara A, Itoh T, Sakai HD, Shimizu M, Yuki M, Kaneko M, Takashina T, Ohkuma M. Nanobdella aerobiophila gen. nov., sp. nov., a thermoacidophilic, obligate ectosymbiotic archaeon, and proposal of Nanobdellaceae fam. nov., Nanobdellales ord. nov. and Nanobdellia class. nov. Int J Syst Evol Microbiol 2022; 72:5489.