白色念珠菌
白色念珠菌(学名:Candida albicans)是一种能造成伺机性感染的酵母菌[5],常见于人类消化道与泌尿生殖道的菌群,约有四成至六成健康成人的口腔与消化道中都有白色念珠菌[6][7],平时与人体行片利共生,但可在人体免疫缺陷[8]时过度生长[7][9]而造成念珠菌症[7][9],是念珠菌属最常见的致病菌种,与热带念珠菌、近平滑念珠菌、光滑念珠菌和克鲁斯念珠菌合计造成了95%的念珠菌症感染[9][10][11],其中白色念珠菌便占了66%,但此比例正持续下降中[12]。白色念珠菌造成的感染有口腔、食管与阴道等处的局部粘膜感染,也有严重的全身性感染,有研究指出后者的致死率高达40%[13],另有一项统计显示白色念珠菌造成的院内感染在美国每年造成2,800至11,200人死亡[14]。白色念珠菌也是最常在医疗器材上形成生物薄膜的真菌,生物薄膜中的白色念珠菌抗性比游离成长者还高,可随着医疗器材入侵血液而造成严重感染[15][16]。
| 白色念珠菌 | |
|---|---|
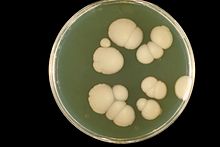
| |
| 生长于沙鲍弱氏琼脂的白色念珠菌 | |
| 科学分类 | |
| 界: | 真菌界 Fungi |
| 门: | 子囊菌门 Ascomycota |
| 纲: | 酵母菌纲 Saccharomycetes |
| 目: | 酵母菌目 Saccharomycetales |
| 科: | 酵母菌科 Saccharomycetaceae |
| 属: | 念珠菌属 Candida |
| 种: | 白色念珠菌 C. albicans
|
| 双名法 | |
| Candida albicans | |
| 异名 | |
白色念珠菌多以双倍体的型式存在,不同于多数真菌为单倍体,但近年渐有研究发现少数情况下白色念珠菌也可形成单倍体与四倍体[17],其双倍体的基因组长度为29Mb[18],另外其基因组中,CUG密码子对应的氨基酸为丝氨酸,与绝大多数生物中对应的亮氨酸不同,是相当罕见的遗传密码例外情形。白色念珠菌常被当作真菌感染研究的模式生物,容易在实验室中培养,并可以改变培养基成分的方式控制其生长型态[19]。白色念珠菌属于双态性真菌,可以酵母型或菌丝型两种方式生长[20],除此之外还可在多种不同的形态表型间互相转换,如白色型-混浊型转换可以改变菌落的外形与基因表达,进而促进准性生殖[21]。
名称与分类
编辑白色念珠菌的学名Candida albicans为一同义反复,其中属名Candida来自拉丁文的candidus,意指白色,种加词albicans来自拉丁文的albicō,意思亦为白色[22]。早在公元前400年,希波克拉底在其著作《论流行病》(Of the Epidemics)中即提到了鹅口疮,可能是白色念珠菌感染的最早记载[2][23]。
白色念珠菌的同物异名有多达上百个[2][24],最早于1853年由法国生物学家查尔斯-菲力毗·罗宾发表描述,命名为Oidium albicans,1890年,德国真菌学家弗里德里希·威廉·措普夫调整其分类,改其学名为Monilia albicans(其中Monilia来自拉丁文的monile,意指项链[22])。1923年,荷兰真菌学家克里斯汀·玛丽·别尔克豪特发表了新属念珠菌属,并将白色念珠菌归入该属,改为现名Candida albicans[25]。
种系发生
编辑已有许多分子系统发生学的研究以不同数量、种类的基因序列分析念珠菌属中各物种的亲缘关系[11][26][27]。念珠菌属中,与白色念珠菌关系最近的物种为都柏林念珠菌(C. dubliniensis),两者组成的演化支与热带念珠菌(C. tropicalis)互为姊妹群,而三者组成的演化支又与近平滑念珠菌(C. parapsilosis)和长孢洛德酵母(Lodderomyces elongisporus)组成的演化支互为姊妹群,以上类群均以双倍体的形式存在,不同于多数真菌为单倍体,显示其共祖可能发生了单倍体-双倍体的转换。这些双倍体的类群与其他单倍体的类群(季也蒙念珠菌、葡萄牙念珠菌与汉逊德巴利酵母)共同组成念珠菌演化支(Candida clade),以上类群均将密码子CUG编码为丝氨酸,而不像绝大多数生物将其编码为亮氨酸,显示念珠菌演化支的共祖可能发生了一次CUG密码子对应的tRNA重组。念珠菌属并非单系群[28],同属念珠菌属的光滑念珠菌不属于念珠菌演化支,而是与酿酒酵母等酵母菌关系较近,共同组成酵母菌演化支(Saccharomyces clade),与其他念珠菌的亲缘关系较远[11]。
| |||||||||||||||||||||||||||||||||||||||||||||||||
基因组
编辑单倍体的白色念珠菌的基因组的大小约为15Mb(双倍体约为29Mb)[29],由8条染色体对组成,二倍体中其中一组染色体分别称为chr1A、chr2A、chr3A、chr4A、chr5A、chr6A、chr7A和chrRA,另一组染色体具有相似的名称,但最后一个字为B,如chr1B、chr2B、...和chrRB。整个基因组包含6198个开放阅读框(ORF),截至2019年2月,这些ORF中仍有70%尚未被鉴定[30]。有研究使用四环素转录调控系统控制白色念珠菌各个基因的表现,建构了一个GRACE(基因置换和条件表达)数据库,研究白色念珠菌基因组中各基因对其生长的必要性,结果显示其中有567个基因是其生长所必须的[31][32]。研究白色念珠菌最常用的菌株是WO-1和SC5314菌株,WO-1菌株可高频率地在白色型(white)与混浊型(opaque)之间互相转换,而SC5314菌株是用作基因组参考序列的菌株[33]。白色念珠菌两种常用菌株的基因组已经完成全基因组测序[8],其中SC5314菌株完成于2004年[34],是较早完成测序的真菌之一。
白色念珠菌核基因组的一大特色是密码子CUG对应的氨基酸不像其他生物中为亮氨酸,而是丝氨酸,同属念珠菌演化支的其他物种亦有此现象[11],这种遗传密码发生例外的情形在真核生物的细胞核基因组中是相当罕见的,在原核生物中也仅出现在起始密码子[35][36][37]。此现象可能使白色念珠菌长期处在热休克反应的状态,有助于提升其对逆境的抗性[38]。有研究分析比较白色念珠菌与酿酒酵母基因中的CUG密码子,发现白色念珠菌的CUG密码子(编码丝氨酸)在酿酒酵母的同源序列中大多是对应编码丝氨酸与其他亲水氨基酸的密码子,而酿酒酵母的CUG密码子(编码亮氨酸)在白色念珠菌的同源序列中大多是对应编码亮氨酸与其他疏水氨基酸的密码子,显示CUG密码子功能的改变并未对蛋白质的构象造成太大变化[11]。有研究将白色念珠菌识别CUG密码子的tRNA导入到酿酒酵母中表现,使其翻译时随机将部分CUG密码子辨认为对应丝氨酸,结果启动了抗逆境反应,造成酵母维持在二倍体的状态,且抑制了交配[39]。非标准的遗传密码使白色念珠菌蛋白质交互作用的研究难以在模式生物酿酒酵母中进行,对此已有研究团队开发了白色念珠菌的双杂合系统[40]。
白色念珠菌的基因组有高度杂合性,有着大量的SNP位点,常因染色体长度多型性(重复序列的长度不一)、相互易位、染色体删除与个别染色体成为三体等因素,造成染色体数量或结构上的变异,诸如此类核型的改变会导致表型的改变,是这种真菌对不同环境适应的机制之一。随着白色念珠菌基因组信息的成功解码,关于这些机制的研究正持续进行中[41][42][43]。
研究工具
编辑白色念珠菌是用来研究真菌感染的重要模式生物[44],许多研究团队开发了白色念珠菌的各种数据库与其分子生物学研究的工具[8]。2004年,多位研究者合作开发了念珠菌基因组数据库(Candida Genome Database, CGD),该数据库是由酵母菌基因组数据库修改而来,许多开发者过去也曾参与酵母菌基因组数据库的构建[45]。念珠菌基因组数据库最初提供白色念珠菌SC5314菌株的基因组信息,以全基因组霰弹枪测序法测序[46]。后来也陆续加入白色念珠菌的其他菌株与念珠菌属的其他物种[45]。
对白色念珠菌进行基因克隆后,常用的选择性标记包括CaNAT1(抗诺尔丝菌素)、MPAr 与IMH3r(抗霉酚酸)等,也有使用营养缺陷型者,例如URA3基因的产物为乳清酸核苷-5'-磷酸脱羧酶,是合成单磷酸尿苷所需的酵素,为白色念珠菌生长所必须,因此URA3营养缺陷型的突变株只有在成功克隆带有URA3的质粒后才能在不含尿嘧啶的特殊培养基中生长,URA3因而可作为基因克隆后的选择性标记,但许多研究显示克隆的URA3从质粒整合进染色体上的位点不同,会使其表现量不同而影响白色念珠菌的致病能力,而可能造成研究中判断克隆基因功能的困难[47],为此已有研究团队开发组氨酸(HIS1)、亮氨酸(LEU2)与精氨酸(ARG4)营养缺陷型的菌株与相应质粒,这些基因的克隆不会影响白色念珠菌的致病能力,以避免使用基因克隆研究特定基因对致病能力影响时,对研究结果造成干扰[48]。
以质粒进行基因克隆时,常用于酿酒酵母的质粒在白色念珠菌中多有在染色体外不稳定或基因表达效率低下的问题,有研究团队开发了适用于白色念珠菌基因克隆的CIp10(Candida integrating plasmid 10)质粒,以核糖体蛋白RP10[注 1]基因为整合进染色体中的同源序列,并带有用作选择性标记的URA3基因[50],因其整合进染色体的位置固定,可避免URA3表现量因整合位点不同而有异,进而影响致病能力[48]。CIp10质粒可以高效率的克隆进白色念珠菌中,并有多个研究团队对其进行微调,设计出各种衍生的质粒[51][52]。
型态
编辑白色念珠菌有许多不同的形态表型,除了作为双态性真菌,可以在酵母菌型态与菌丝型态间转换之外,还可以进行通称为“高频率转换”(high-frequency switching)之多种其他表型间的转换,其中被研究最多的一种转换则是某些菌株白色型-混浊型间的转换[53],另外还有许多其他不同的转换系统,例如SC5314菌株可在七种表型间互相转换[54][55]。酵母菌型-菌丝型转换的过程相当快速,且通常整个菌落的所有细胞都会一起转换;高频率转换则可能只在菌落中的部分细胞中发生,且经转换后的细胞通常仍能进行酵母菌型-菌丝型转换。这些转换均为可逆,且常受到二氧化碳浓度、氧气浓度、培养基种类与温度等环境因子的影响[56]。
酵母菌型─菌丝型转换
编辑白色念珠菌为双态性真菌,可以酵母菌或菌丝的型式生长,其中酵母菌型生长时可进行出芽生殖,有时出芽产生的子细胞没有立即脱离,而形成类似菌丝的丝状外观,称为假菌丝(pseudohypha)[57]。白色念珠菌在实验室常用的标准培养液生长时通常呈酵母菌型,呈卵圆形,长约6微米[20],不过温度、二氧化碳浓度、养分与酸碱值的些许改变便可能使其转换成菌丝型[58][59],在模拟人体生理环境的培养基中生长时亦呈菌丝型。在适合其生长的环境下,白色念珠菌菌丝萌发时直径约为2.6微米,成熟菌丝直径则约为3.4微米[20]。两种型态在白色念珠菌感染人体时各有功能,酵母菌型较适合在血液中散播,菌丝型则有助于穿透组织、在器官中着生、躲避巨噬细胞的攻击、以及在医疗器材上形成生物薄膜[57][60][61],这种型态转换与其致病力密切相关,不过仍有研究指出有无法进行此转换的菌株亦可进行感染。以小分子药物抑制白色念珠菌的酵母菌型─菌丝型转换可能有助于抑制其感染。[62][63]
另外白色念珠菌还在环境条件恶劣时,可以形成对逆境抗性较高的厚垣孢子。厚垣孢子多着生于从菌丝分枝出去之瓶状突起的端点[64]。
高频率转换
编辑除了酵母菌型-菌丝型转换外,白色念珠菌亦可进行高频率转换(high-frequency switching),在多种表型间相互转换,这种转换常是自然发生,并非由特定环境因子调控,且只发生在菌落中的部分细胞[56]。高频率转换最早于1985年由美国生物学家大卫·索尔的团队在SC5314菌株中发现,这种菌株可以在平滑(smooth)[注 2]、星状(star)、不规则皱纹状(irregular wrinkle)、环状(ring)、点状(stipple)、帽状(hat)与绒毛状(fuzzy)等七种表型间进行转换,不同表型间转换发生的频率各异,介于万分之一至十分之一之间,且紫外线的照射可大幅提升转换发生的频率[53][55]。高频率转换可能与某些基因表达的改变有关,有研究发现缺乏组蛋白脱乙酰酶SIR2的菌株出现高频率转换的几率大幅上升,显示控制转换的基因平时可能为SIR2所抑制[65]。在各种表型间转换的能力有助于白色念珠菌在不同环境下的生长[66]。
白色型─混浊型转换
编辑白色型─混浊型转换(white-to-opaque switching)是白色念珠菌第二种被发现的高频率转换,于1986年由大卫·索尔团队在WO-1菌株中发现,其中白色型(white)的细胞呈圆形,形成白色、平滑的菌落,混浊型(opaque)的细胞形状则较长,形成灰色且较为扁平的菌落[54]。此转换是由WOR1(White to Opaque Regulator 1)蛋白调控,WOR1蛋白则受MTL基因调控,当MTLa与MTLα基因同时存在时,会产生a1-α2复合体,抑制WOR1蛋白的表现,进而抑制白色型─混浊型的转换[67],又因混浊型是发生准性生殖的主要表型,进行准性生殖的几率比白色型高上百万倍[68][69],自然环境中有高达95%的白色念珠菌都属MTLa/α型,因此难有此转换与交配发生[21][68]。
白色型─混浊型转换受到许多环境因子调控,其中氧化压力会促进白色念珠菌转为混浊型,温度的提升则会促进其转为白色型,因此在宿主体内(摄氏37度)生长时白色念珠菌多为白色型,但缺氧[注 3]、二氧化碳与N-乙酰葡糖胺[注 4]浓度高的环境(例如消化道中)可以亦可刺激白色型─混浊型转换,使白色念珠菌呈混浊型,因此感染宿主的白色念珠菌亦可能发生交配[71]。另外当白色念珠菌生长速率降低时,可能因细胞中得以累积足够浓度的WOR1蛋白,不因细胞分裂而快速被稀释,进而促进白色型─混浊型转换[68]。白色型─混浊型转换对白色念珠菌的致病力也有关联。白色型较适于体内的全身性感染,混浊型则较适于皮肤的浅表感染,两种型态对宿主免疫系统的反应与耐受性也有所不同,可能有助于感染[68]。
除了白色念珠菌外,同为念珠菌属且亦可感染人体的都柏林念珠菌与热带念珠菌也能进行白色型─混浊型转换[72]。2014年,有研究发现除了白色型与混浊型外,还有一种“灰色型”可与两型态相互转换,灰色型的白色念珠菌造成皮肤感染的能力比另外两种型态还强,发生交配的几率则介于两者之间,且白色型─灰色型的转换不需WOR1的参与。三种型态各有不同的基因表达,造成其型态、代谢、致病能力、适应宿主体内的环境与对逆境的抗性皆有所不同[72]。
其他转换
编辑除了白色型─混浊型转换外,还有许多新的白色念珠菌表型转换机制陆续被发现,分别存在不同菌株中[56]。从人体阴道中分离的部分菌株可以在菌丝量多与菌丝量少的菌落型态之间互相转换[73]。SC5314菌株还可进行白色型─消化道型的转换,消化道型(Gastrointestinally-IndUced Transition, GUT)是存在宿主消化道中的类型,其外型与混浊型相似,细胞形状较长、形成的菌落较扁,且从白色型转换的过程亦需WOR1蛋白参与,但以电子显微镜观察时可见其表面没有混浊型常有的突起,且基因表达亦与混浊型不同,并因维持MTLa/α的基因型而不像混浊型可以进行交配。消化道中可能含有刺激白色型白色念珠菌转换为消化道型的讯息,此型的白色念珠菌主要在宿主消化道中行片利共生,不对宿主健康造成负面影响[74][75]。
生殖
编辑白色念珠菌过去被认为只能以有丝分裂进行无性生殖,不能进行减数分裂和产生孢子,没有以有性生殖繁殖的途径。现代研究则发现白色念珠菌亦可进行准性生殖[76],但仅限于MTLa或MTLα基因型的个体,MTLa/α者因会抑制白色型-混浊型转换而难以交配(详见#白色型─混浊型转换一节)[21]。混浊型的MTLa与MTLα的个体可分别释放MFa与MFα费洛蒙,与彼此细胞表面的受体结合而启动停止细胞周期、改变细胞型态等交配机制[77],并可形成一种称为性菌膜(sexual biofilm)的生物薄膜,有助于交配进行[76][78]。最终两细胞可进行核聚变而形成四倍体。四倍体在某些环境下不稳定,有丝分裂时可能发生染色体不分离而产生非整倍体,进而在多次有丝分裂后回复为二倍体[79],其中染色体缺失的过程有协同效应,即丢失数个染色体后,在后续的有丝分裂中继续丢失其他染色体的几率会提升[80]。有丝分裂时Spo11蛋白可以切割DNA,促使同源染色体间发生同源重组[76][79]。
白色念珠菌因为双倍体,其准性生殖为二倍体-四倍体-二倍体的过程,而不同于其他真菌的单倍体-二倍体-单倍体。不过白色念珠菌的二倍体亦可能丢失染色体而形成单倍体,单倍体的a与α菌株亦可交配而形成二倍体[76][81]。
致病机制
编辑白色念珠菌可以分泌天冬氨酸蛋白酶(secreted aspartic proteinases, Saps)、磷脂酶与脂酶,可分解宿主细胞的胞外底物与细胞膜表面物质[82],有助其黏着、侵入宿主的组织中[83],甚至可能可以用以破坏免疫细胞[82]。2016年,有研究发现白色念珠菌感染宿主粘膜时,从酵母菌型转为菌丝型后,会分泌一种称为念珠菌素的毒素,念珠菌素长31个氨基酸,是由Kex2p蛋白酶切割Ece1p产生的一种多肽,可造成宿主上皮细胞的损伤,并刺激宿主的免疫反应[84]。
酵母菌型-菌丝型转换对白色念珠菌的致病能力相当重要。吞噬细胞是宿主抗真菌免疫反应中的重要环节,白色念珠菌被巨噬细胞内吞后,并由酵母菌型转换为菌丝型,最终穿透巨噬细胞而重新回到组织中。在巨噬细胞内部,白色念珠菌会启动许多基因的表现,改变其代谢与促进型态转换,其中许多基因为念珠菌所特有,酿酒酵母中不存在同源基因者,很可能与白色念珠菌的致病能力有关[85]。另外菌丝型的白色念珠菌菌丝表面有Hwp1(Hyphal wall protein 1)蛋白,该蛋白可与宿主上皮细胞表面的转麸酰胺酶结合,有助白色念珠菌黏附于宿主细胞,进而造成感染[86]。
生物薄膜
编辑白色念珠菌还可形成结构复杂的生物薄膜,由圆球形的酵母菌、假菌丝与菌丝等各种型态的细胞与胞外底物组成,其基因表达与游离的细胞有很大的不同,其中Efg1、Tec1、Bcr1、Ndt80、Brg1与Rob1等六个转录因子对生物薄膜的形成特别重要,另外可能有上千个基因都对生物薄膜的形成有所影响[87][88]。生物薄膜可以多种机制提高白色念珠菌对抗真菌剂的抗性,包括增加外排泵的表现以将药物排出胞外,以胞外底物中的水解酶分解药物,以及形成抗性很强的休眠细胞(persister cells)。生物薄膜中的酵母菌还可以向外传播,这些细胞比起正常的酵母菌型细胞有较强的黏附力,形成新的生物薄膜的能力较强,具有较高的致病能力[88]。
白色念珠菌在环境中与人体中皆可形成生物薄膜。在医疗器材上,白色念珠菌是最常被发现的真菌种类[15],导尿管、静脉导管、心律调节器、人工瓣膜、人工关节、隐形眼镜与假牙等医疗器材上均可能有白色念珠菌的生物薄膜生长。生物薄膜中的白色念珠菌较正常游离者对人体免疫系统的抗性较高,可能借由医疗器材入侵循环系统,造成严重的全身性感染,还可能使这些医疗器材无法正常运作[88]。
在人体中,白色念珠菌还可能与许多细菌共同形成生物薄膜,包括口腔中的转糖链球菌、格氏链球菌、粘性放线菌或细梭菌,肠道中的松脆杆菌、产气荚膜梭菌、大肠杆菌、克雷伯氏肺炎菌与粪肠球菌,阴道中的乳杆菌属细菌与囊肿纤维症患者肺中的绿脓杆菌,白色念珠菌与这些细菌可透过实际接触、分泌讯息分子或改变环境因子来影响彼此的行为,例如绿脓杆菌可分泌一种内酯,抑制生物薄膜中白色念珠菌菌丝的形成,但不影响其整体生长速率[88][89]。还有研究显示白色念珠菌形成的生物薄膜提供了缺氧的微环境,使松脆杆菌与产气荚膜梭菌等厌氧细菌得以生长,且这些细菌可能可以促进白色念珠菌形成生物薄膜,以利自己在有氧环境下生长。白色念珠菌与细菌共同形成的生物薄膜对人体健康的影响仍不清楚[88]。
感染
编辑白色念珠菌为伺机性感染的致病真菌,平时可生长于人体的皮肤与粘膜组织中而不造成负面影响,但当其过度生长时可能造成念珠菌症的感染。念珠菌症又可依感染部位分为若干类型,包括感染口部的鹅口疮、感染阴道的念珠菌性外阴阴道炎、感染食管的念珠菌性食管炎等,甚至可能造成全身的侵入性感染,侵犯多个器官,并造成念珠菌血症(Candidemia)[90]。念珠菌症在健康成人中比较少见,小于一个月的新生儿、艾滋病患者、癌症患者、糖尿病患者与服用抗生素与皮质类固醇等免疫缺乏者感染的风险较高[91]。白色念珠菌也常造成医疗照顾相关感染[92]。
浅表与局部感染
编辑口腔感染
编辑据统计,世界上有一半的人群口腔中有白色念珠菌[6],甚至有更精密的方法显示90%的人皆有,但绝大多数不造成任何感染症状[93],免疫缺乏、高糖分的饮食、配戴假牙、抽烟与口干皆会造成口腔中白色念珠菌出现的几率增加[94]。若白色念珠菌造成口腔粘膜损伤即为鹅口疮,通常不伴随其他症状且没有痛感,只有部分患者会有灼痛感[95],其他可能的症状还包括味觉改变、触感改变、患部发红等[91]。医护人员常透过简单观察口腔即作出鹅口疮的诊断,有时则需取小量检体进行培养,再于显微镜下观察鉴别。治疗鹅口疮使用的抗真菌剂包括克催玛汝、咪康唑与耐丝菌素,较严重者则可能施以氟康唑[91]。
食管感染
编辑念珠菌性食管炎为白色念珠菌造成的食管感染,多发生于艾滋病患者、接受化疗的癌症患者等免疫缺乏的人群,但有时亦见于免疫力正常者中。念珠菌性食管炎的症状包括疼痛与吞咽困难,其诊断多为使用内视镜,治疗多使用氟康唑。感染念珠菌性食管炎的患者通常同时患有口腔感染[91]。
阴道感染
编辑白色念珠菌为两成女性阴道菌丛的正常成分,并不造成感染,若因激素、服用药物或免疫力下降导致阴道环境改变,而使白色念珠菌大量增殖,便可能导致念珠菌性外阴阴道炎。免疫缺乏、服用避孕药、服用抗生素与妊娠均会增加感染机会,症状包括阴道痛痒、性交与排尿时有痛感与异常的分泌物,诊断多需取小量样本进行培养,再于显微镜下观察鉴别。治疗方式于阴道涂抹抗真菌剂或口服氟康唑,严重者除继续口服氟康唑外,还可能于患部施以硼酸、耐丝菌素或氟胞嘧啶[96]。
念珠菌性外阴阴道炎是第二常见的阴道炎,仅次于细菌性阴道病[97]。有高达75%的女性在其一生中得过至少一次念珠菌性外阴阴道炎,接近半数超过两次[98][99]。约5%的女性在一年中被感染三次以上[99]。
其他
编辑除了以上部位之外,白色念珠菌还可感染指甲而造成念珠菌性甲癣[100],感染甲沟造成念珠菌性甲沟炎[101],感染嘴角造成口角炎[102],感染舌头导致正中菱形舌炎[102],感染男性生殖器导致龟头炎[103],感染毛囊导致毛囊炎[103],在消化道过度生长而造成肠胃不适等[104]。产道存有白色念珠菌的孕妇若发生早期羊膜囊破裂,可能造成新生儿感染先天性皮肤念珠菌病[105]。
全身性感染
编辑白色念珠菌亦可造成全身性的感染,侵入血液、心脏、眼、骨骼等部位,其中最常见者为念珠菌血症(Candidemia),为常见的医疗照顾相关感染,在美国每年约有46000例[106],患者多为原本就患有其他重大疾病者[107],高风险者包括装置静脉导管的患者、住院期间长的患者、加护病房的患者、施用广效型抗生素的患者、烧烫伤患者、接受全静脉营养治疗者与嗜中性白细胞低下者[92]。
全身性念珠菌症通常是以血液样本培养进行诊断,多数患者最初治疗的推荐用药为棘白菌素,感染的菌株种类与抗药性确认后则改为氟康唑,部分患者也会以氟康唑为起始用药。成功将血液中的白色念珠菌清除并消除症状后,疗程还需持续两周[108]。1999年,一篇发表于《Clinical Infectious Diseases》的研究统计美国49间医院3年间所有在院内发生的血液感染,共记录了超过一万起感染,其中念珠菌属真菌为第四常见的病原,且以白色念珠菌为大宗[57][109]。念珠菌血症的死亡率可高达40%-60%[8][110],每年造成的医疗花费超过十亿美元[57]。
注脚
编辑参考资料
编辑- ^ Candida albicans at NCBI Taxonomy browser (页面存档备份,存于互联网档案馆)
- ^ 2.0 2.1 2.2 Kurtzman, C. P.; Fell, Jack W. The yeasts, a taxonomic study 4. 1998. ISBN 978-0444813121.
- ^ Simi, Vincent. Origin of the Names of Species of Candida (PDF). [2018-12-06]. (原始内容存档 (PDF)于2015-06-21).
- ^ McClary, Dan Otho. Factors Affecting the Morphology of Candida Albicans. Annals of the Missouri Botanical Garden. May 1952, 39 (2): 137–164. JSTOR 2394509. doi:10.2307/2394509.
- ^ Gow, N.A.R. Microbe Profile: Candida albicans: a shape-changing, opportunistic pathogenic fungus of humans. Microbiology. 2017, 163 (8): 1145–1147. PMID 28809155. doi:10.1099/mic.0.000499.
- ^ 6.0 6.1 Kerawala C, Newlands C (编). Oral and maxillofacial surgery. Oxford: Oxford University Press. 2010: 446, 447. ISBN 978-0-19-920483-0.
- ^ 7.0 7.1 7.2 Erdogan A, Rao SS. Small intestinal fungal overgrowth. Curr Gastroenterol Rep. April 2015, 17 (4): 16. PMID 25786900. doi:10.1007/s11894-015-0436-2.
- ^ 8.0 8.1 8.2 8.3 Calderone A, Clancy CJ (编). Candida and Candidiasis 2nd. ASM Press. 2012. ISBN 978-1-55581-539-4.
- ^ 9.0 9.1 9.2 Martins N, Ferreira IC, Barros L, Silva S, Henriques M. Candidiasis: predisposing factors, prevention, diagnosis and alternative treatment. Mycopathologia. June 2014, 177 (5–6): 223–240. PMID 24789109. doi:10.1007/s11046-014-9749-1.
- ^ Pfaller MA, Diekema DJ. Epidemiology of invasive candidiasis: a persistent public health problem. Clin Microbiol Rev. January 2007, 20 (1): 133–163. PMC 1797637 . PMID 17223626. doi:10.1128/CMR.00029-06.
- ^ 11.0 11.1 11.2 11.3 11.4 Geraldine Butler, Matthew D. Rasmussen, Christina A. Cuomo. Evolution of pathogenicity and sexual reproduction in eight Candida genomes. Nature. 2009, 459 (7247): 657-662. doi:10.1038/nature08064.
- ^ M. A. Pfaller, D. J. Diekema. Epidemiology of Invasive Candidiasis: a Persistent Public Health Problem. Clinical Microbiology Reviews. 2007, 20 (1): 133-163. doi:10.1128/CMR.00029-06.
- ^ Singh, Rachna; Chakrabarti, Arunaloke. Invasive Candidiasis in the Southeast-Asian Region. Prasad, Rajendra (编). Candida albicans: Cellular and Molecular Biology 2. Switzerland: Springer International Publishing AG. 2017: 27. ISBN 978-3-319-50408-7.
- ^ Pfaller, M. A.; Diekema, D. J. Epidemiology of Invasive Candidiasis: A Persistent Public Health Problem. Clinical Microbiology Reviews. 2007, 20 (1): 133–63. PMC 1797637 . PMID 17223626. doi:10.1128/CMR.00029-06.
- ^ 15.0 15.1 Kumamoto CA. Candida biofilms. Current Opinion in Microbiology. 2002, 5 (6): 608–11. PMID 12457706. doi:10.1016/s1369-5274(02)00371-5.
- ^ Donlan RM. Biofilm formation: a clinically relevant microbiological process. Clinical Infectious Diseases. 2001, 33 (8): 1387–92. PMID 11565080. doi:10.1086/322972.
- ^ Hickman MA, Zeng G, Forche A, Hirasawa MP, Abbey D, Harrison BD, Wang YM, Su CH, Bennett RJ, Wang Y, Berman J. The 'obligate diploid' Candida albicans forms mating-competent haploids. Nature. 2016, 494 (7435): 55–59. Bibcode:2013Natur.494...55H. PMC 3583542 . PMID 23364695. doi:10.1038/nature11865.
- ^ Candida albicans SC5314 Genome Snapshot/Overview. www.candidagenome.org. [2018-03-27]. (原始内容存档于2018-11-16).
- ^ Development of Candida albicans Hyphae in Different Growth Media – Variations in Growth Rates, Cell Dimensions and Timing of Morphogenetic Events (PDF) (132). 1986.
- ^ 20.0 20.1 20.2 Frans M. Klis, Chris G. de Koster, Stanley Brul. Cell Wall-Related Bionumbers and Bioestimates of Saccharomyces cerevisiae and Candida albicans. Eukaryot Cell. 2014, 13 (1): 2–9. doi:10.1128/EC.00250-13.
- ^ 21.0 21.1 21.2 Christina M. Hull, Ryan M. Raisner, Alexander D. Johnson. Evidence for Mating of the "Asexual" Yeast Candida albicans in a Mammalian Host. Science. 2000, 289 (5477): 307-310. doi:10.1126/science.289.5477.307.
- ^ 22.0 22.1 William S. Haubrich. Medical Meanings: A Glossary of Word Origins. ACP Press. 2003: 39 [2019-02-22]. ISBN 1930513496. (原始内容存档于2021-11-02).
- ^ McCool, Logan. The Discovery and Naming of Candida albicans (PDF). antimicrobe.org. [2019-02-22]. (原始内容存档 (PDF)于2018-05-05).
- ^ Candida albicans. MycoBank. [2019-02-22]. (原始内容存档于2019-02-22).
- ^ Simi, Vincent. Origin of the Names of Species of Candida (PDF). antimicrobe.org. [2018-12-06]. (原始内容存档 (PDF)于2015-06-21).
- ^ Sung-Oui Suh, Meredith Blackwell, Cletus P. Kurtzman, Marc-André Lachance. Phylogenetics of Saccharomycetales, the ascomycete yeasts. Mycologia. 2006, 98 (6): 1006-1017. doi:10.1080/15572536.2006.11832629.
- ^ Stephanie Diezmann, Cymon J. Cox, Gabriele Schönian, Rytas J. Vilgalys, Thomas G. Mitchell. Phylogeny and Evolution of Medical Species of Candida and Related Taxa: a Multigenic Analysis. Journal of Clinical Microbiology. 2004, 42 (12): 5624–5635. doi:10.1128/JCM.42.12.5624-5635.2004.
- ^ Stefanie Mühlhausen, Martin Kollmar. Molecular Phylogeny of Sequenced Saccharomycetes Reveals Polyphyly of the Alternative Yeast Codon Usage. Genome Biol Evol. 2014, 6 (12): 3222–3237. doi:10.1093/gbe/evu152.
- ^ Ted Jones, Nancy A. Federspiel, Hiroji Chibana, Jan Dungan, Sue Kalman, B. B. Magee, George Newport,Yvonne R. Thorstenson, Nina Agabian, P. T. Magee, Ronald W. Davis, Stewart Scherer. The diploid genome sequence of Candida albicans. PNAS. 2004, 101 (19): 7329–7334. doi:10.1073/pnas.0401648101.
- ^ Skrzypek MS, Binkley J, Binkley G, Miyasato SR, Simison M, and Sherlock G. Candida albicans SC5314 Genome Snapshot/Overview. Candida Genome Database. 2019-02-19 [2018-12-06]. (原始内容存档于2018-11-16).
- ^ Roemer T, Jiang B, Davison J, Ketela T, Veillette K, Breton A, Tandia F, Linteau A, Sillaots S, Marta C, Martel N, Veronneau S, Lemieux S, Kauffman S, Becker J, Storms R, Boone C, Bussey H. Large-scale essential gene identification in Candida albicans and applications to antifungal drug discovery. Mol Microbiol. 2003, 38 (19): 167–81. PMID 14507372. doi:10.1046/j.1365-2958.2003.03697.x.
- ^ Candida Community News. www.candidagenome.org. [2018-03-27]. (原始内容存档于2018-10-27).
- ^ Candida Strains. www.candidagenome.org. [2018-03-27]. (原始内容存档于2018-10-27).
- ^ Jones T, Federspiel NA, Chibana H, Dungan J, Kalman S, Magee BB, Newport G, Thorstenson YR, Agabian N, Magee PT, Davis RW, Scherer S. The diploid genome sequence of Candida albicans. Proc Natl Acad Sci U S A. 2004, 101 (19): 7329–7334. PMC 409918 . PMID 15123810. doi:10.1073/pnas.0401648101.
- ^ Ohama, T; Suzuki, Tsutomu; Mori, Miki; Osawa, Syozo; Ueda, Takuya; Watanabe, Kimitsuna; Nakase, Takashi. Non-universal decoding of the leucine codon CUG in several Candida species. Nucleic Acids Research. August 1993, 21 (17): 1039–4045. PMC 309997 . PMID 8371978. doi:10.1093/nar/21.17.4039.
- ^ Arnaud, MB; Costanzo, MC; Inglis, DO; Skrzypek, MS; Binkley, J; Shah, P; Binkley, G; Miyasato, SR; Sherlock, G. CGD Help: Non-standard Genetic Codes. Candida Genome Database. [2011-10-30]. (原始内容存档于2018-11-01).
- ^ Andrzej (Anjay) Elzanowski and Jim Ostell. The Alternative Yeast Nuclear Code. The Genetic Codes. Bethesda, Maryland, U.S.A.: National Center for Biotechnology Information (NCBI). 2010-07-07 [2011-10-30]. (原始内容存档于2011-05-13).
- ^ Santos, MA; Cheesman, C; Costa, V; Moradas-Ferreira, P; Tuite, MF. Selective advantages created by codon ambiguity allowed for the evolution of an alternative genetic code in Candida spp.. Molecular Microbiology. February 1999, 31 (3): 937–947. PMID 10048036. doi:10.1046/j.1365-2958.1999.01233.x.
- ^ Raquel M Silva, João A Paredes, Gabriela R Moura, Bruno Manadas, Tatiana Lima‐Costa, Rita Rocha, Isabel Miranda, Ana C Gomes, Marian JG Koerkamp, Michel Perrot, Frank CP Holstege, Hélian Boucherie, Manuel A S Santos. Critical roles for a genetic code alteration in the evolution of the genus Candida. EMBO J. October 2007, 26 (21): 4555–65. PMC 2063480 . PMID 17932489. doi:10.1038/sj.emboj.7601876.
- ^ Stynen, B; Van Dijck, P; Tournu, H. A CUG codon adapted two-hybrid system for the pathogenic fungus Candida albicans. Nucleic Acids Res. October 2010, 38 (19): e184. PMC 2965261 . PMID 20719741. doi:10.1093/nar/gkq725.
- ^ Rustchenko-Bulgac, E. P. Variations of Candida albicans Electrophoretic Karyotypes. J. Bacteriol. 1991, 173 (20): 6586–6596. PMC 208996 . PMID 1917880. doi:10.1128/jb.173.20.6586-6596.1991.
- ^ Holmes, Ann R.; Tsao, Sarah; Ong, Soo-Wee; Lamping, Erwin; Niimi, Kyoko; Monk, Brian C.; Niimi, Masakazu; Kaneko, Aki; Holland, Barbara R.; Schmid, Jan; Cannon, Richard D. Heterozygosity and functional allelic variation in the Candida albicans efflux pump genes CDR1 and CDR2. Molecular Microbiology. 2006, 62 (1): 170–86. PMID 16942600. doi:10.1111/j.1365-2958.2006.05357.x.
- ^ Jones, T.; Federspiel, N. A.; Chibana, H.; Dungan, J.; Kalman, S.; Magee, B. B.; Newport, G.; Thorstenson, Y. R.; Agabian, N.; Magee, P. T.; Davis, R. W.; Scherer, S. The diploid genome sequence of Candida albicans. Proceedings of the National Academy of Sciences. 2004, 101 (19): 7329–7334. Bibcode:2004PNAS..101.7329J. PMC 409918 . PMID 15123810. doi:10.1073/pnas.0401648101.
- ^ Maria C. Costanzo, Martha B. Arnaud, Marek S. Skrzypek, Gail Binkley, Christopher Lane, Stuart R. Miyasato, Gavin Sherlock. The Candida Genome Database: Facilitating research on Candida albicans molecular biology. FEMS Yeast Research. 2006, 6 (5): 671-684. doi:10.1111/j.1567-1364.2006.00074.x.
- ^ 45.0 45.1 Marek S. Skrzypek, Jonathan Binkley, Gavin Sherlock. Using the Candida Genome Database. Methods Mol Biol: 31–47. doi:10.1007/978-1-4939-7737-6_3.
- ^ van het Hoog, Marco; Rast, Timothy J; Martchenko, Mikhail; Grindle, Suzanne; Dignard, Daniel; Hogues, Hervé; Cuomo, Christine; Berriman, Matthew; Scherer, Stewart; Magee, BB; Whiteway, Malcolm; Chibana, Hiroji; Nantel, André; Magee, PT. Assembly of the Candida albicans genome into sixteen supercontigs aligned on the eight chromosomes. Genome Biology. 2007, 8 (4): R52. PMC 1896002 . PMID 17419877. doi:10.1186/gb-2007-8-4-r52.
- ^ Cheng, S; Nguyen, M. H; Zhang, Z; Jia, H; Handfield, M; Clancy, C. J. Evaluation of the Roles of Four Candida albicans Genes in Virulence by Using Gene Disruption Strains That Express URA3 from the Native Locus. Infection and Immunity. 2003, 71 (10): 6101–3. PMC 201070 . PMID 14500538. doi:10.1128/IAI.71.10.6101-6103.2003.
- ^ 48.0 48.1 Noble, S. M; Johnson, A. D. Strains and strategies for large-scale gene deletion studies of the diploid human fungal pathogen Candida albicans. Eukaryotic Cell. 2005, 4 (2): 298–309. PMC 549318 . PMID 15701792. doi:10.1128/EC.4.2.298-309.2005.
- ^ Swoboda RK, Broadbent ID, Bertram G, Budge S, Gooday GW, Gow NA, Brown AJ. Structure and regulation of a Candida albicans RP10 gene which encodes an immunogenic protein homologous to Saccharomyces cerevisiae ribosomal protein 10. J Bacteriol. 1995, 177 (5): 1239-1246. doi:10.1128/jb.177.5.1239-1246.1995.
- ^ Abdul Munir Abdul Murad, Philip R. Lee, Ian D. Broadbent, Caroline J. Barelle, Alistair J. P. Brown. CIp10, an efficient and convenient integrating vector for Candida albicans. Yeast. 2000, 14 (4): 325-327. doi:10.1002/1097-0061(20000315)16:4<325::AID-YEA538>3.0.CO;2-%23.
- ^ Chauvel, Murielle; Nesseir, Audrey; Cabral, Vitor; Znaidi, Sadri; Goyard, Sophie; Bachellier-Bassi, Sophie; Firon, Arnaud; Legrand, Mélanie; Diogo, Dorothée; Naulleau, Claire; Rossignol, Tristan; d'Enfert, Christophe. A Versatile Overexpression Strategy in the Pathogenic Yeast Candida albicans: Identification of Regulators of Morphogenesis and Fitness. PLOS One. 2012, 7 (9): e45912. Bibcode:2012PLoSO...745912C. PMC 3457969 . PMID 23049891. doi:10.1371/journal.pone.0045912.
- ^ Walker, Louise A.; MacCallum, Donna M.; Bertram, Gwyneth; Gow, Neil A.R.; Odds, Frank C.; Brown, Alistair J.P. Genome-wide analysis of Candida albicans gene expression patterns during infection of the mammalian kidney. Fungal Genetics and Biology. 2009, 46 (2): 210–9. PMC 2698078 . PMID 19032986. doi:10.1016/j.fgb.2008.10.012.
- ^ 53.0 53.1 Neena Jain, Fahmi Hasan, Bettina C. Fries. Phenotypic Switching in Fungi. Curr Fungal Infect Rep. 2008, 2 (3). doi:10.1007/s12281-008-0026-y.
- ^ 54.0 54.1 Slutsky, B; Staebell, M; Anderson, J; Risen, L; Pfaller, M; Soll, DR. "White-opaque transition": a second high-frequency switching system in Candida albicans.. J. Bacteriol. 1987, 1 (169): 189–197. PMC 211752 . PMID 3539914. doi:10.1128/jb.169.1.189-197.1987.
- ^ 55.0 55.1 55.2 Slutsky, B; Buffo, J; Soll, D. R. High-frequency switching of colony morphology in Candida albicans. Science. 1985, 230 (4726): 666–9. Bibcode:1985Sci...230..666S. PMID 3901258. doi:10.1126/science.3901258.
- ^ 56.0 56.1 56.2 Soll, DR. High-frequency switching in Candida albicans. Clin Microbiol Rev. 1992, 5 (2): 183–203. PMC 358234 . PMID 1576587. doi:10.1128/cmr.5.2.183.
- ^ 57.0 57.1 57.2 57.3 Sudbery, P; Gow, N; Berman, J. The distinct morphogenic states of Candida albicans. Trends in Microbiology. 2004, 12 (7): 317–24. PMID 15223059. doi:10.1016/j.tim.2004.05.008.
- ^ Si H, Hernday AD, Hirasawa MP, Johnson AD, Bennett RJ. Candida albicans White and Opaque Cells Undergo Distinct Programs of Filamentous Growth. PLoS Pathog. 2013, 9 (3): e1003210. PMC 3591317 . PMID 23505370. doi:10.1371/journal.ppat.1003210.
- ^ Peter E. Sudbery. Growth of Candida albicans hyphae (PDF). Nature Reviews Microbiology. 2011, 9 (10): 737–748. PMID 21844880. doi:10.1038/nrmicro2636.。参见figure 2 (页面存档备份,存于互联网档案馆)
- ^ Jiménez-López, Claudia; Lorenz, Michael C. Fungal Immune Evasion in a Model Host–Pathogen Interaction: Candida albicans Versus Macrophages. PLoS Pathogens. 2013, 9 (11): e1003741. PMC 3836912 . PMID 24278014. doi:10.1371/journal.ppat.1003741.
- ^ Berman J, Sudbery PE. Candida Albicans: a molecular revolution built on lessons from budding yeast. Nature Reviews Genetics. 2002, 3 (12): 918–930. PMID 12459722. doi:10.1038/nrg948.
- ^ Shareck, J.; Belhumeur, P. Modulation of Morphogenesis in Candida albicans by Various Small Molecules. Eukaryotic Cell. 2011, 10 (8): 1004–12. PMC 3165445 . PMID 21642508. doi:10.1128/EC.05030-11.
- ^ Suzanne M Noble, Sarah French, Lisa A Kohn, Victoria Chen, Alexander D Johnson. Systematic screens of a Candida albicanshomozygous deletion library decouple morphogenetic switching and pathogenicity. Nature Genetics. 2010, 42 (7): 590–598. doi:10.1038/ng.605.
- ^ Peter Staib, Joachim Morschhäuser. Chlamydospore formation in Candida albicans and Candida dubliniensis– an enigmatic developmental programme. Mycoses. 2006, 50 (1): 1-12. doi:10.1111/j.1439-0507.2006.01308.x.
- ^ José Pérez‐Martín, José Antonio Uría, Alexander D. Johnson. Phenotypic switching in Candida albicans is controlled by a SIR2 gene. The EMBO Journal. 1999, 18 (9): 2580-2592. doi:10.1093/emboj/18.9.2580.
- ^ Tao L, Du H, Guan G, Dai Y, Nobile C, Liang W, Cao C, Zhang Q, Zhong J, Huang G. Discovery of a "White-Gray-Opaque" Tristable Phenotypic Switching System in Candida albicans: Roles of Non-genetic Diversity in Host Adaptation. PLoS Biol. 2014, 12 (4): e1001830. PMC 3972085 . PMID 24691005. doi:10.1371/journal.pbio.1001830.
- ^ Morschhäuser J. Regulation of white-opaque switching in Candida albicans. Med Microbiol Immunol.. 2010, 199 (3): 165–172. PMID 20390300. doi:10.1007/s00430-010-0147-0.
- ^ 68.0 68.1 68.2 68.3 Lohse MB, Johnson AD. White-opaque switching in Candida albicans. Curr Opin Microbiol. 2009, 12 (6): 650–654. PMC 2812476 . PMID 19853498. doi:10.1016/j.mib.2009.09.010.
- ^ Hnisz D, Tscherner M, Kuchler K. Opaque-white phenotype transition: a programmed morphological transition in Candida albicans. Methods Mol. Biol. Methods in Molecular Biology. 2011, 734 (2): 303–315. ISBN 978-1-61779-085-0. PMID 21468996. doi:10.1007/978-1-61779-086-7_15.
- ^ Bernardo Ramírez-Zavala, Oliver Reuß, Yang-Nim Park, Knut Ohlsen,Joachim Morschhäuser. Environmental Induction of White–Opaque Switching in Candida albicans. PLoS Pathogens. 2008, 4 (6): e1000089. doi:10.1371/journal.ppat.1000089.
- ^ 71.0 71.1 Guanghua Huang, Song Yi, Nidhi Sahni, Karla J. Daniels, Thyagarajan Srikantha, David R. Soll. N-Acetylglucosamine Induces White to Opaque Switching, a Mating Prerequisite in Candida albicans. PLoS Pathogens. 2010, 6 (3): e1000806. doi:10.1371/journal.ppat.1000806.
- ^ 72.0 72.1 Li Tao, Han Du, Guobo Guan, Yu Dai, Clarissa J. Nobile, Weihong Liang, Chengjun Cao, Qiuyu Zhang, Jin Zhong, Guanghua Huang. Discovery of a “White-Gray-Opaque” Tristable Phenotypic Switching System in Candida albicans: Roles of Non-genetic Diversity in Host Adaptation. PLoS Biology. 2014, 12 (4): e1001830. doi:10.1371/journal.pbio.1001830.
- ^ D R Soll, C J Langtimm, J McDowell, J Hicks, R Galask. High-frequency switching in Candida strains isolated from vaginitis patients. Journal of Clinical Microbiology. 1987, 25 (9): 1611-1622 [2019-03-01]. (原始内容存档于2019-03-01).
- ^ Pande, Kalyan; Chen, Changbin; Noble, Suzanne M. Passage through the mammalian gut triggers a phenotypic switch that promotes Candida albicans commensalism. Nature Genetics. 2013, 45 (9): 1088–91. PMC 3758371 . PMID 23892606. doi:10.1038/ng.2710.
- ^ Noble, Suzanne M.; Gianetti, Brittany A.; Witchley, Jessica N. Candida albicans cell-type switching and functional plasticity in the mammalian host. Nature Reviews Microbiology. 2016, 15 (2): 96–108. PMC 5957277 . PMID 27867199. doi:10.1038/nrmicro.2016.157.
- ^ 76.0 76.1 76.2 76.3 Richard J Bennett. The parasexual lifestyle of Candida albicans. Current Opinion in Microbiology. 2015, 28: 10-17. doi:10.1016/j.mib.2015.06.017.
- ^ Kevin Alby, Richard J. Bennett. Sexual reproduction in the Candida clade: cryptic cycles, diverse mechanisms, and alternative functions. Cell Mol Life Sci.. 2010, 67 (19): 3275–3285. doi:10.1007/s00018-010-0421-8.
- ^ Yang-Nim Park, Karla J. Daniels, Claude Pujol, Thyagarajan Srikantha, David R. Soll. Candida albicans Forms a Specialized “Sexual” as Well as “Pathogenic” Biofilm. Eukaryotic Cell. 2013, 12 (8): 1120-1131. doi:10.1128/EC.00112-13.
- ^ 79.0 79.1 Anja Forche, Kevin Alby, Dana Schaefer, Alexander D Johnson, Judith Berman, Richard J Bennett. The Parasexual Cycle in Candida albicans Provides an Alternative Pathway to Meiosis for the Formation of Recombinant Strains. PLoS Biology. 2008, 6 (5): e110. doi:10.1371/journal.pbio.0060110.
- ^ Richard J. Bennett, Alexander D. Johnson. Completion of a parasexual cycle in Candida albicans by induced chromosome loss in tetraploid strains. The EMBO Journal: 2505-2515. doi:10.1093/emboj/cdg235.
- ^ Meleah A. Hickman, Guisheng Zeng, Anja Forche, Matthew P. Hirakawa, Darren Abbey, Benjamin D. Harrison, Yan-Ming Wang, Ching-hua Su, Richard J. Bennett, Yue Wang, Judith Berman. The ‘obligate diploid’ Candida albicansforms mating-competent haploids. Nature. 2013, 494 (7435): 55-59. doi:10.1038/nature11865.
- ^ 82.0 82.1 Schaller M, Borelli C, Korting HC, Hube B. Hydrolytic enzymes as virulence factors of Candida albicans. Mycoses. 2005, 48 (6): 365-377. doi:10.1111/j.1439-0507.2005.01165.x.
- ^ Monika S, Małgorzata B, Zbigniew O. Contribution of Aspartic Proteases in Candida Virulence. Protease Inhibitors against Candida Infections. Curr Protein Pept Sci.. 2017, 18 (10): 1050-1062. doi:10.2174/1389203717666160809155749.
- ^ Duncan Wilson; Julian R. Naglik; Bernhard Hube. The Missing Link between Candida albicans Hyphal Morphogenesis and Host Cell Damage. PLoS Pathog. 2016, 12 (10): e1005867. PMC 5072684 . PMID 27764260. doi:10.1371/journal.ppat.1005867.
- ^ Lorenz, M. C; Bender, J. A; Fink, G. R. Transcriptional Response of Candida albicans upon Internalization by Macrophages. Eukaryotic Cell. 2004, 3 (5): 1076–87. PMC 522606 . PMID 15470236. doi:10.1128/EC.3.5.1076-1087.2004.
- ^ Staab, J. F. Adhesive and Mammalian Transglutaminase Substrate Properties of Candida albicans Hwp1. Science. 1999, 283 (5407): 1535–1538. Bibcode:1999Sci...283.1535S. ISSN 0036-8075. doi:10.1126/science.283.5407.1535.
- ^ Clarissa J. Nobile, Emily P.Fox, Jeniel E.Nett, Trevor R. Sorrells, Quinn M. Mitrovich, Aaron D. Hernday, Brian B. Tuch, David R.Andes, Alexander D. Johnson. A Recently Evolved Transcriptional Network Controls Biofilm Development in Candida albicans. Cell. 2012, 148 (1-2): 126-138. doi:10.1016/j.cell.2011.10.048.
- ^ 88.0 88.1 88.2 88.3 88.4 Clarissa J. Nobile and Alexander D. Johnson. Candida albicans Biofilms and Human Disease. Annu Rev Microbiol.. 2015, 69: 71-92. doi:10.1146/annurev-micro-091014-104330.
- ^ Deborah A. Hogan, Åshild Vik, Roberto Kolter. A Pseudomonas aeruginosa quorum‐sensing molecule influences Candida albicans morphology. Molecular Microbiology. 2004, 54 (5): 1212=1223. doi:10.1111/j.1365-2958.2004.04349.x.
- ^ Invasive Candidiasis. Centers for Disease Control and Prevention. 2015-06-12 [2019-03-01]. (原始内容存档于2019-01-09).
- ^ 91.0 91.1 91.2 91.3 Candida infections of the mouth, throat, and esophagus. Centers for Disease Control and Prevention. 2017-08-04 [2019-03-01]. (原始内容存档于2019-01-09).
- ^ 92.0 92.1 Emily R. M. Sydnor, Trish M. Perl. Hospital Epidemiology and Infection Control in Acute-Care Settings. Clinical Microbioly Review. 2011, 24 (1): 141–173. doi:10.1128/CMR.00027-10.
- ^ Greenberg MS, Glick M, Ship JA. Burket's oral medicine 11th. Hamilton, Ont.: BC Decker. 2008: 79–84. ISBN 9781550093452.
- ^ Scully C. Oral and maxillofacial medicine : the basis of diagnosis and treatment 3rd. Edinburgh: Churchill Livingstone. 2013: 254–267. ISBN 9780702049484.
- ^ Rhodus, NL. Treatment of oral candidiasis (PDF). Northwest Dentistry. Mar–Apr 2012, 91 (2): 32–3 [2019-03-01]. PMID 22662470. (原始内容 (PDF)存档于2013-11-02).
- ^ Vaginal Candidiasis. Centers for Disease Control and Prevention. 2017-08-04 [2019-03-01]. (原始内容存档于2014-12-29).
- ^ Ilkit, M; Guzel, AB. The epidemiology, pathogenesis, and diagnosis of vulvovaginal candidosis: a mycological perspective. Critical Reviews in Microbiology. August 2011, 37 (3): 250–61. PMID 21599498. doi:10.3109/1040841X.2011.576332.
- ^ Vaginal yeast infections fact sheet. womenshealth.gov. 2014-12-23 [2015-03-05]. (原始内容存档于2015-03-04).
- ^ 99.0 99.1 Egan ME, Lipsky MS. Diagnosis of vaginitis. Am Fam Physician. September 2000, 62 (5): 1095–104. PMID 10997533. (原始内容存档于2011-06-06).
- ^ James, William D.; Berger, Timothy G. Andrews' Diseases of the Skin: clinical Dermatology. Saunders Elsevier. 2006: 305. ISBN 0-7216-2921-0.
- ^ James, William D.; Berger, Timothy G. Andrews' Diseases of the Skin: clinical Dermatology. Saunders Elsevier. 2006: 310. ISBN 978-0-7216-2921-6.
- ^ 102.0 102.1 Patil S, Rao RS, Majumdar B, Anil S. Clinical Appearance of Oral Candida Infection and Therapeutic Strategies. Front. Microbiol. December 2015, 6: 1391. PMC 4681845 . PMID 26733948. doi:10.3389/fmicb.2015.01391.
- ^ 103.0 103.1 Hidalgo JA, Vazquez JA. Candidiasis: Clinical Presentation. Medscape. WebMD. 2015-08-18 [2016-06-22]. (原始内容存档于2016-06-01).
- ^ Wang ZK, Yang YS, Stefka AT, Sun G, Peng LH. Review article: fungal microbiota and digestive diseases. Aliment. Pharmacol. Ther. April 2014, 39 (8): 751–766. PMID 24612332. doi:10.1111/apt.12665.
- ^ James, William D.; Berger, Timothy G. Andrews' Diseases of the Skin: clinical Dermatology. Saunders Elsevier. 2006: 309. ISBN 0-7216-2921-0.
- ^ Invasive Candidiasis Statistics. Centers for Disease Control and Prevention. 2017-01-25 [2019-03-01]. (原始内容存档于2014-12-29).
- ^ Symptoms of Invasive Candidiasis. Centers for Disease Control and Prevention. 2015-06-12 [2019-03-01]. (原始内容存档于2014-12-29).
- ^ Information for Healthcare Professionals about Invasive Candidiasis. Centers for Disease Control and Prevention. 2015-06-12 [2019-03-01]. (原始内容存档于2019-01-09).
- ^ Michael B. Edmond, Sarah E. Wallace, Donna K. McClish, Michael A. Pfaller, Ronald N. Jones, and Richard P. Wenzel. Nosocomial Bloodstream Infections in United States Hospitals: A Three-Year Analysis. Clinical Infectious Diseases. 1999, 29 (2): 239–244. doi:10.1086/520192.
- ^ Weinberger, M. Characteristics of candidaemia with Candida-albicans compared with non-albicans Candida species and predictors of mortality. J Hosp Infect. 2016-04-16, 61 (2): 146–54. PMID 16009456. doi:10.1016/j.jhin.2005.02.009.